同じく信用はしないでね。
これでやらかした間違いは、非線形なのに線形として計算しちゃったこと。「先週から今週で50%になったので、日々50/7%減ったのだ」としちゃダメなわけよ、バカでしたわ。ワイドショーがやってたのは「先週から今週で50%になったので、来週さらに50%」としてきっちり一週間後の予測をしようとしてただけで、そこに至る曲線には無頓着だった、ので、ワイドショーがやってたやり方そのものは正しい。単にワタシがアホだっただけ。
んでは:
1 # -*- coding: utf-8 -*-
2 import io
3 import sys
4 import csv
5 import datetime
6 import numpy as np
7
8
9 def _tonpa(fn):
10 # rownum=2020/1/16からの日数, colnum=都道府県コード-1
11 result = {
12 "newpat": np.zeros((1, 47)),
13 "newpatmovavg": np.zeros((1, 47)), # 週平均
14 "newpatmovavg2": np.zeros((1, 47)), # 2週平均
15 "newdeath": np.zeros((1, 47)),
16 "newdeathmovavg": np.zeros((1, 47)),
17 "newdeathmovavg2": np.zeros((1, 47)),
18 "effrepro": np.zeros((1, 48)), # 実効再生産数(47都道府県+全国)
19 }
20 era = "2020/1/16"
21 era = datetime.datetime(*map(int, era.split("/")))
22 reader = csv.reader(io.open(fn, encoding="utf-8-sig"))
23 next(reader)
24 for row in reader:
25 dt, (p, c, d) = row[0], map(int, row[1::2])
26 dt = (datetime.datetime(*map(int, dt.split("/"))) - era).days
27 if dt > len(result["newpat"]) - 1:
28 for k in result.keys():
29 cn = 48 if k == "effrepro" else 47
30 result[k] = np.vstack((result[k], np.zeros((1, cn))))
31 result["newpat"][dt, p - 1] = c
32 result["newpatmovavg"][dt, p - 1] = result[
33 "newpat"][max(0, dt - 7):dt + 1, p - 1].mean()
34 result["newpatmovavg2"][dt, p - 1] = result[
35 "newpat"][max(0, dt - 14):dt + 1, p - 1].mean()
36 result["newdeath"][dt, p - 1] = d
37 result["newdeathmovavg"][dt, p - 1] = result[
38 "newdeath"][max(0, dt - 7):dt + 1, p - 1].mean()
39 result["newdeathmovavg2"][dt, p - 1] = result[
40 "newdeath"][max(0, dt - 14):dt + 1, p - 1].mean()
41 # 実効再生産数は、現京都大学西浦教授提案による簡易計算(を一般向け説明にまとめてくれ
42 # ている東洋経済ONLINE)より:
43 # (直近7日間の新規陽性者数/その前7日間の新規陽性者数)^(平均世代時間/報告間隔)
44 # 平均世代時間は=5日、報告間隔=7日の仮定。
45 # のつもりだが、東洋経済ONLINEで GitHub にて公開(閉鎖予定)されてる計算結果の csv とは
46 # 値が違う。違うことそのものは致し方ないこと。計算タイミングによって結果は変わるし、
47 # NHK まとめと東洋経済ONLINEのデータはシンクロしてないから。思いっきり間違ってるって
48 # ことはなさそう。近い値にはなってる。
49 for dt in range(len(result["newpat"])):
50 # 都道府県別
51 sum1 = result["newpat"][dt-6:dt+1, :47].sum(axis=0)
52 sum2 = result["newpat"][dt-6-7:dt+1-7, :47].sum(axis=0)
53 result["effrepro"][dt, :47] = np.power(sum1 / sum2, 5. / 7.)
54
55 # 全国
56 sum1 = result["newpat"][dt-6:dt+1, :].sum()
57 sum2 = result["newpat"][dt-6-7:dt+1-7, :].sum()
58 result["effrepro"][dt, 47] = np.power(sum1 / sum2, 5. / 7.)
59 np.savez(io.open("nhk_news_covid19_prefectures_daily_data.npz", "wb"), **result)
60
61
62 if __name__ == '__main__':
63 fn = "nhk_news_covid19_prefectures_daily_data.csv"
64 _tonpa(fn)
スクリプトをマジメに読んだ人は気付いたかもしれない。「8日平均」と「15日平均」を計算しちゃってる。一連の「主張」の本題とはあまり関係ないとはいえ、さすがにもうちょっと慎重にやるべきでした、すまぬ。正しくは「dt – 6」「dt – 5」…「dt – 1」「dt」の7エレメントでの平均を取らなければならないので、「slice(dt – 6, dt + 1)」。8日平均が役に立たないわけではないけれど、やりたいこととやってることが違うので、間違いは間違い。グラフは滑らかさが若干減って、もうほんの少しだけガタガタになる。
1 # -*- coding: utf-8 -*-
2 import io
3 import datetime
4 import numpy as np
5 from scipy.optimize import curve_fit
6 import matplotlib as mpl
7 import matplotlib.pyplot as plt
8 import matplotlib.font_manager
9
10
11 def _vis(dat):
12 fontprop = matplotlib.font_manager.FontProperties(
13 fname="c:/Windows/Fonts/meiryo.ttc")
14
15 era = "2020/1/16" # 木曜日
16 era = datetime.datetime(*map(int, era.split("/")))
17 (wh_sl, wh_n) = (slice(12, 12+1), "東京")
18
19 npat = dat["newpat"][:,wh_sl].sum(axis=1)
20 npatma = dat["newpatmovavg"][:,wh_sl].sum(axis=1)
21
22 T = [era + datetime.timedelta(t) for t in range(len(npat))]
23 fig, ax1 = plt.subplots(tight_layout=True)
24 fig.set_size_inches(16.53 * 1.2, 11.69)
25 skip = 7*4*10
26 #
27 # ワイドショーが最近やってる「単純計算でこのまま~%減少ペースならば云々」そのもの
28 # を。簡単のために直近の最大(1月13日)と本日の比較とする。計算は要は「a^t * n0」の
29 # a を求める問題。
30 #
31 n0 = npatma[skip:].max()
32 [t0], = np.where(npatma[skip:] == n0) # 1月13日
33 def _fcurve(t, a):
34 return n0 * np.power(a, t)
35 t1 = len(npatma[skip:]) - 1
36 popt, pcov = curve_fit(_fcurve, np.arange(t1 - t0 + 1), npatma[skip:][t0:])
37 preds = []
38 val = npatma[skip:][t1]
39 while True:
40 preds.append(val)
41 if val < 500.: # 東京の感染者数週平均が500を下回る?
42 break
43 val *= popt[0]
44 #
45 lines = []
46 l, = ax1.plot(T[skip:], npatma[skip:])
47 lines.append(l)
48 l, = ax1.plot(
49 [era + datetime.timedelta(skip + t)
50 for t in range(t1, t1 + len(preds))], preds)
51 lines.append(l)
52 ax1.legend(
53 lines,
54 ["7日移動平均実績", "減少率{:.2f}(per days)での週平均予測".format(popt[0])],
55 shadow=True, prop=fontprop)
56 ax1.set_xlabel(
57 "{}の新規感染者数推移\n(「NHK新型コロナウイルス関連データ」を加工)".format(
58 wh_n),
59 fontproperties=fontprop)
60 plt.axhspan(0, 500, facecolor="lightgray", alpha=0.1)
61 fig.autofmt_xdate()
62 #
63 tlast = era + datetime.timedelta(skip + t1 + len(preds) - 1)
64 for evtxt, evdt in (
65 ("GoToトラベル全国一時停止", datetime.datetime(2020, 12, 15)),
66 ("1都3県に緊急事態宣言", datetime.datetime(2021, 1, 7)),
67 ("緊急事態宣言11都府県に拡大", datetime.datetime(2021, 1, 13)),
68 ("東京の週平均が500を下回る?", tlast),
69 ):
70 ax1.annotate("{} ({})".format(evtxt, str(evdt)[:len("????-??-??")]),
71 xy=(evdt, 0), xycoords='data',
72 xytext=(
73 0.5,
74 (evdt - era).days / (tlast - era).days),
75 textcoords='axes fraction',
76 arrowprops=dict(facecolor='black', width=0.0001, alpha=0.1),
77 horizontalalignment='right', verticalalignment='top',
78 alpha=0.3,
79 fontproperties=fontprop)
80 ax1.vlines([evdt], 0, 1, transform=ax1.get_xaxis_transform(), alpha=0.1)
81 #
82 #plt.show()
83 fig.savefig(
84 "nhk_news_covid19_prefectures_daily_data5.png")
85 plt.close(fig)
86
87
88 if __name__ == '__main__':
89 fn = "nhk_news_covid19_prefectures_daily_data.npz"
90 _vis(np.load(io.open(fn, "rb")))
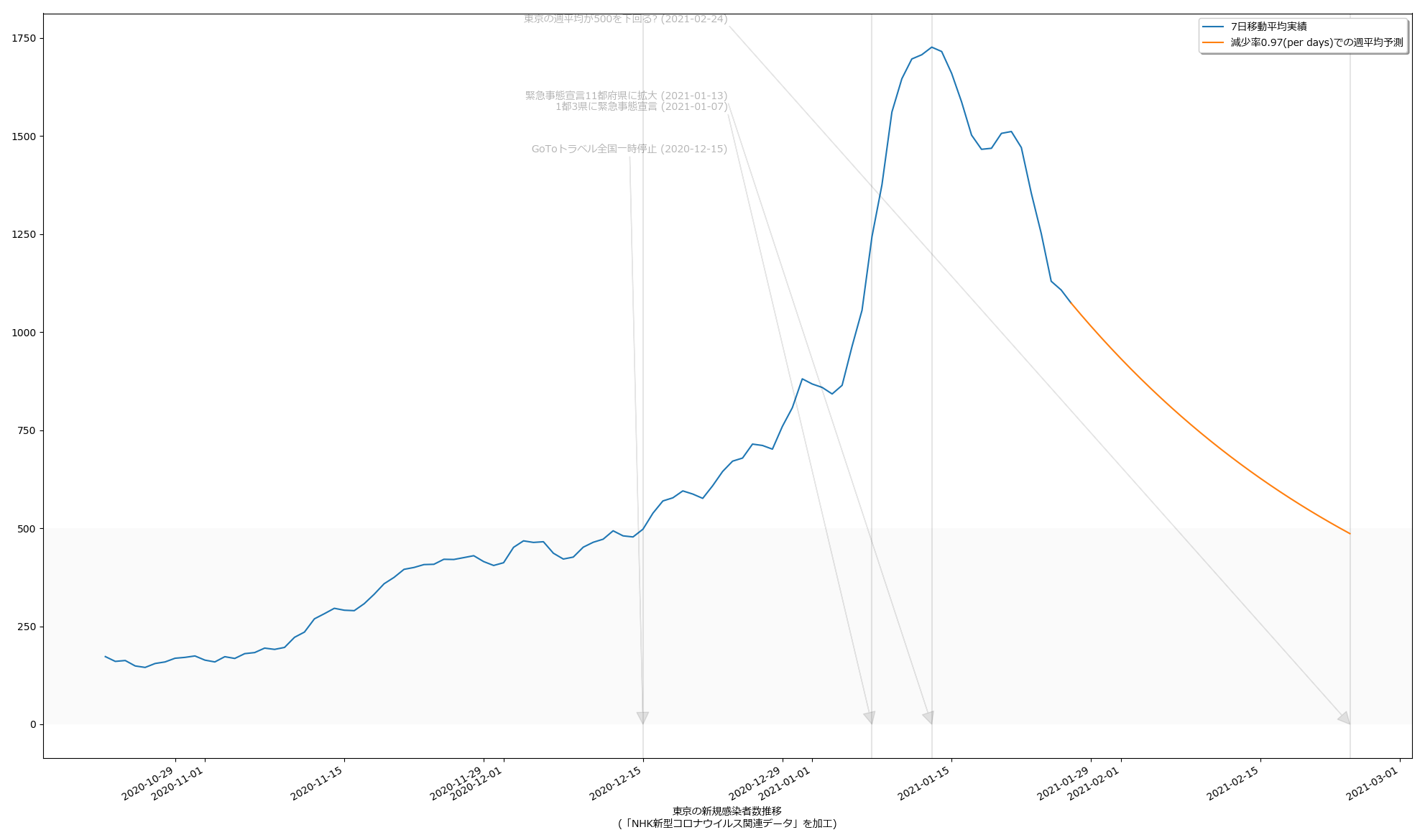
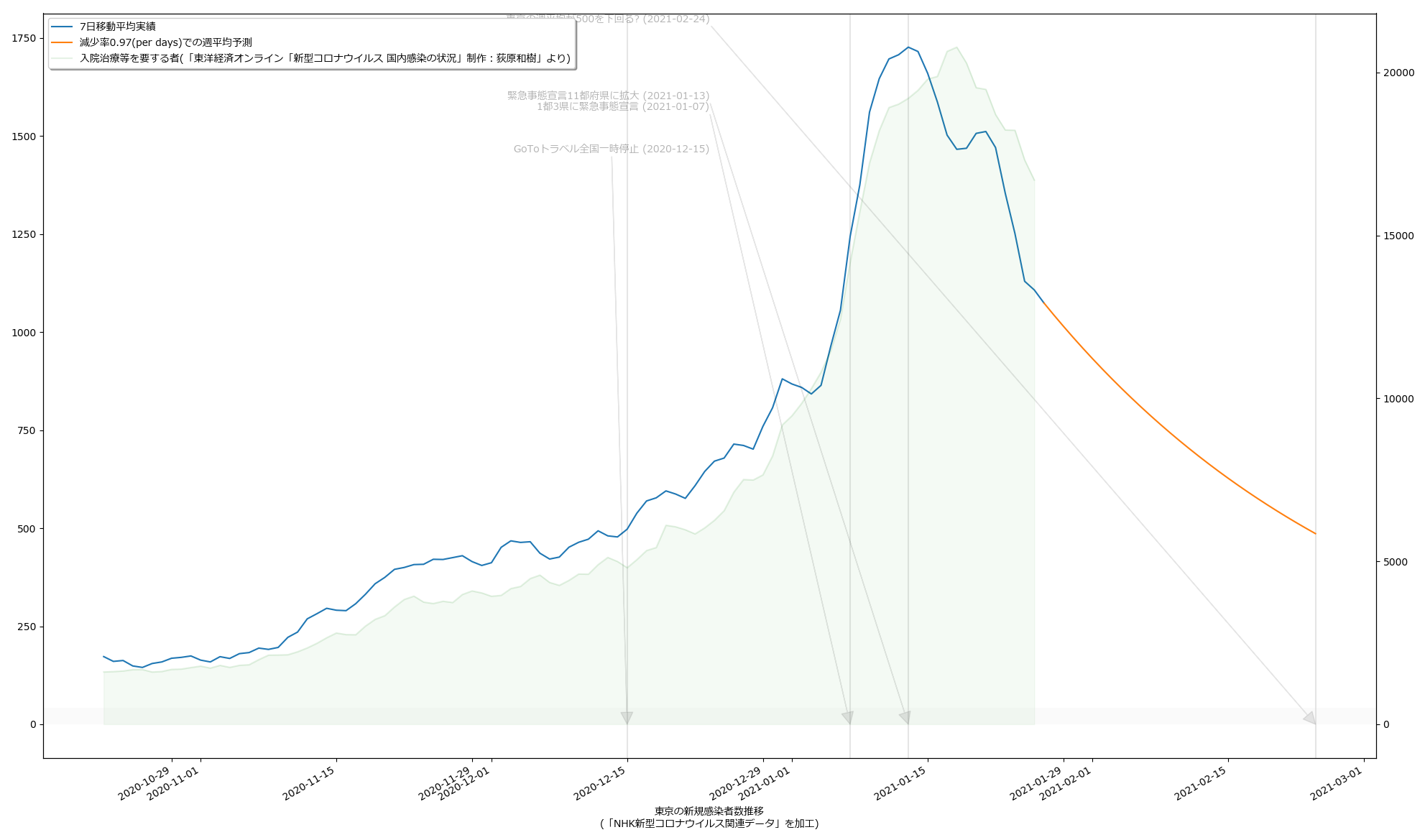
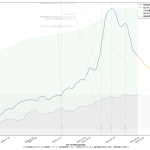
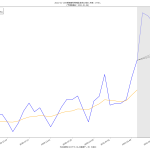
毎日3%ずつ減っていけば2月24日には500を下回る、って計算ね。うん、第一印象でなんとなく感じてたのってこんなくらいだった。違和感はあんまりない。でもグラフの傾きをみてると、最善の予測ではやはり二月中旬にも下回るという予測になってても、それはそれで違和感はない。
どっちにしても、まずは2月7日という線は「まぁ絶望的だろう」、そして、2月末までなのか、中旬なのか、てとこだよね。あと、(8)の追記の方で試した、1%しか減らないという最悪のシナリオだとこれはとんでもないことになるので、そうはならないことを願うばかり。
2021-01-28朝追記:
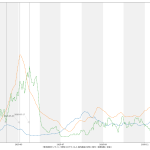
「総合的に=なんとなく」とならないために本来真っ先に必要になるのが近未来予測であり、その予測において最も重要な数値が「医療逼迫度」なのであろう、ということなのであれば、ここまででやってきた新規陽性者予測だけでなく病床使用率予測をすべきであろう、てことなのだけれども、少なくとも一般人がオープンなデータだけに基づいてこれを求めるのは結構酷で、少なくとも NHK まとめのデータは必要データが不足しすぎている。
東洋経済ONLINEのものが NHK のものより種類が多いが、これはこれで見せ方が今一つ不十分なのよね。解析に必要なデータという意味ではまぁまぁでも、何かを主張するための見方には足らず、そうしたければ「解析の必要がある」。
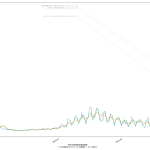
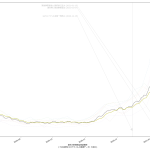
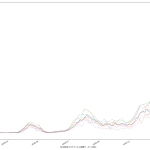
なんかね、ちょっととてつもなく「惜しい」んだと思うんだよなぁ。「入院治療等を要する者」なんだけど、これ、言い回しは非常に紛らわしくて、理解するまで結構時間かかってしまったんだが、要するに「現時点で感染者扱いになってる人数」てこと。具体的には「現在の入院・宿泊療養・自宅療養者数合計」。データを自分でダウンロードしてくれば手には入るんだけど、可視化されてるグラフでは、なぜか「入院治療等を要する者」の都道府県別がない。もったいない。
「医療逼迫度」を見える化したいとする場合、実際のところ「入院治療等を要する者」の可視化だけでは不十分で、ここに「入院・宿泊療養・自宅療養の区分」や即応病床確保数も同時に可視化する必要がある。けれどもそれをする元ネタは NHK まとめにも 東洋経済ONLINEまとめにもない。そうなのだが、ただ、一般人がおおよそでも状況を把握するのに、「「入院治療等を要する者」の可視化」が常にみえるようになってるのは、悪くないことだと思う。
(いつもにも増して未整理なので)スクリプトは割愛するけれど、「入院治療等を要する者」の実数を一緒にプロットするとこんな具合: