「見える化」がブームとなり、だがそれ以前に報道というものはプレゼンというものを職業にしているわけなのだから、…、なのに、てことよ。
日々のニュース・ワイドショーをみてて、巨大パネルに命をかけてるわりには「肝心の見える化には無頓着なのね」って感じるわけなのよね。「感覚に訴えかける」のに可視化以上のものってないよな、と、改めて理解したよ:
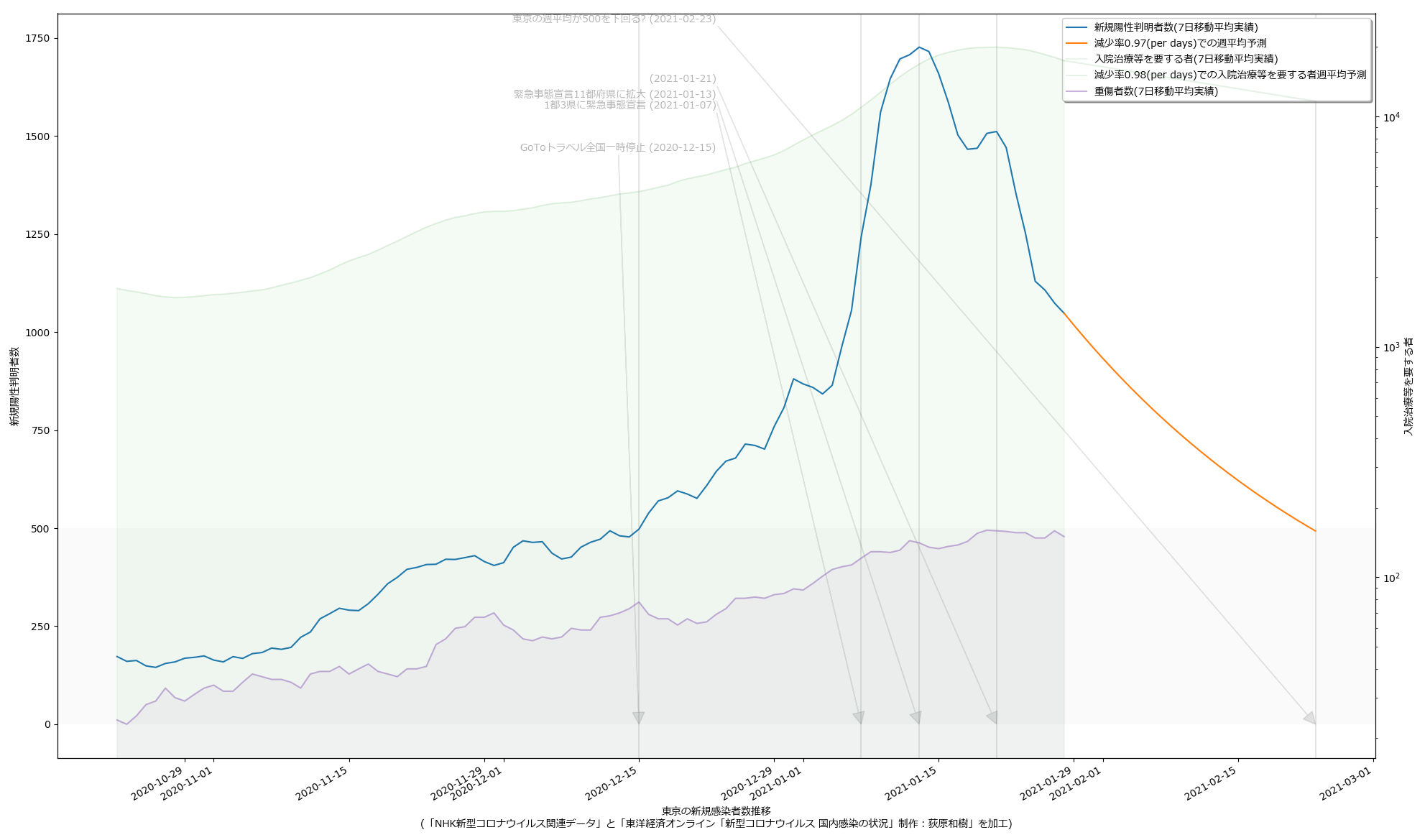
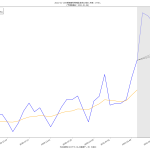
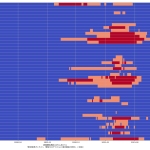
そう、重症者数に関しては「まったく減少傾向の気配がない」。言葉では伝えてるよね。そういうのをさ、「わかりやすくプレゼンする」のって、テレビの十八番なんではないのか、って思わん? こういう図を日々見せられれば、さすがに市民や「バカ議員」の意識も多少は変わると思うんだよね。
いつもスクリプトを見せてるので今回も一応:
1 # -*- coding: utf-8 -*-
2 import io
3 import sys
4 import csv
5 import datetime
6 import numpy as np
7
8
9 def _tonpa(fn):
10 # rownum=2020/1/16からの日数, colnum=都道府県コード-1
11 result = {
12 "newpat": np.zeros((1, 47)),
13 "newpatmovavg": np.zeros((1, 47)), # 週平均
14 "newpatmovavg2": np.zeros((1, 47)), # 2週平均
15 "newdeath": np.zeros((1, 47)),
16 "newdeathmovavg": np.zeros((1, 47)),
17 "newdeathmovavg2": np.zeros((1, 47)),
18 "effrepro": np.zeros((1, 48)), # 実効再生産数(47都道府県+全国)
19 }
20 era = "2020/1/16"
21 era = datetime.datetime(*map(int, era.split("/")))
22 reader = csv.reader(io.open(fn, encoding="utf-8-sig"))
23 next(reader)
24 for row in reader:
25 dt, (p, c, d) = row[0], map(int, row[1::2])
26 dt = (datetime.datetime(*map(int, dt.split("/"))) - era).days
27 if dt > len(result["newpat"]) - 1:
28 for k in result.keys():
29 cn = 48 if k == "effrepro" else 47
30 result[k] = np.vstack((result[k], np.zeros((1, cn))))
31 result["newpat"][dt, p - 1] = c
32 result["newpatmovavg"][dt, p - 1] = result[
33 "newpat"][max(0, dt - 7):dt + 1, p - 1].mean()
34 result["newpatmovavg2"][dt, p - 1] = result[
35 "newpat"][max(0, dt - 14):dt + 1, p - 1].mean()
36 result["newdeath"][dt, p - 1] = d
37 result["newdeathmovavg"][dt, p - 1] = result[
38 "newdeath"][max(0, dt - 7):dt + 1, p - 1].mean()
39 result["newdeathmovavg2"][dt, p - 1] = result[
40 "newdeath"][max(0, dt - 14):dt + 1, p - 1].mean()
41 # 実効再生産数は、現京都大学西浦教授提案による簡易計算(を一般向け説明にまとめてくれ
42 # ている東洋経済ONLINE)より:
43 # (直近7日間の新規陽性者数/その前7日間の新規陽性者数)^(平均世代時間/報告間隔)
44 # 平均世代時間は=5日、報告間隔=7日の仮定。
45 # のつもりだが、東洋経済ONLINEで GitHub にて公開(閉鎖予定)されてる計算結果の csv とは
46 # 値が違う。違うことそのものは致し方ないこと。計算タイミングによって結果は変わるし、
47 # NHK まとめと東洋経済ONLINEのデータはシンクロしてないから。思いっきり間違ってるって
48 # ことはなさそう。近い値にはなってる。
49 for dt in range(len(result["newpat"])):
50 # 都道府県別
51 sum1 = result["newpat"][dt-6:dt+1, :47].sum(axis=0)
52 sum2 = result["newpat"][dt-6-7:dt+1-7, :47].sum(axis=0)
53 result["effrepro"][dt, :47] = np.power(sum1 / sum2, 5. / 7.)
54
55 # 全国
56 sum1 = result["newpat"][dt-6:dt+1, :].sum()
57 sum2 = result["newpat"][dt-6-7:dt+1-7, :].sum()
58 result["effrepro"][dt, 47] = np.power(sum1 / sum2, 5. / 7.)
59 np.savez(io.open("nhk_news_covid19_prefectures_daily_data.npz", "wb"), **result)
60
61
62 if __name__ == '__main__':
63 fn = "nhk_news_covid19_prefectures_daily_data.csv"
64 _tonpa(fn)
スクリプトをマジメに読んだ人は気付いたかもしれない。「8日平均」と「15日平均」を計算しちゃってる。一連の「主張」の本題とはあまり関係ないとはいえ、さすがにもうちょっと慎重にやるべきでした、すまぬ。正しくは「dt – 6」「dt – 5」…「dt – 1」「dt」の7エレメントでの平均を取らなければならないので、「slice(dt – 6, dt + 1)」。8日平均が役に立たないわけではないけれど、やりたいこととやってることが違うので、間違いは間違い。グラフは滑らかさが若干減って、もうほんの少しだけガタガタになる。
1 # -*- coding: utf-8-unix -*-
2 import io
3 import csv
4 import datetime
5 import numpy as np
6
7
8 _PREFCODE = {
9 '北海道': 1,
10 '青森県': 2,
11 '岩手県': 3,
12 '宮城県': 4,
13 '秋田県': 5,
14 '山形県': 6,
15 '福島県': 7,
16 '茨城県': 8,
17 '栃木県': 9,
18 '群馬県': 10,
19 '埼玉県': 11,
20 '千葉県': 12,
21 '東京都': 13,
22 '神奈川県': 14,
23 '新潟県': 15,
24 '富山県': 16,
25 '石川県': 17,
26 '福井県': 18,
27 '山梨県': 19,
28 '長野県': 20,
29 '岐阜県': 21,
30 '静岡県': 22,
31 '愛知県': 23,
32 '三重県': 24,
33 '滋賀県': 25,
34 '京都府': 26,
35 '大阪府': 27,
36 '兵庫県': 28,
37 '奈良県': 29,
38 '和歌山県': 30,
39 '鳥取県': 31,
40 '島根県': 32,
41 '岡山県': 33,
42 '広島県': 34,
43 '山口県': 35,
44 '徳島県': 36,
45 '香川県': 37,
46 '愛媛県': 38,
47 '高知県': 39,
48 '福岡県': 40,
49 '佐賀県': 41,
50 '長崎県': 42,
51 '熊本県': 43,
52 '大分県': 44,
53 '宮崎県': 45,
54 '鹿児島県': 46,
55 '沖縄県': 47,
56 }
57 _COLSNM = (
58 "testedPositive",
59 "peopleTested",
60 "hospitalized",
61 "serious",
62 "discharged",
63 "deaths",
64 "effectiveReproductionNumber",
65
66 "hospitalized_movavg",
67 "serious_movavg",
68 )
69 def _tonpa(fn):
70 # 元データ prefectures.csv の著作権:
71 # 『東洋経済オンライン「新型コロナウイルス 国内感染の状況」制作:荻原和樹』
72 #
73 # 1. 開始日が NHK まとめと違う
74 # 2. 更新のない都道府県は省略してある
75 # 3. 日付は一応抜けなく連続している、今のところ
76 #
77 # NHKまとめのほうで密行列として扱った同じノリにしたい、ここでは面倒だけど。
78 # つまり、列方向に都道府県、行方向に日付。
79 result = {
80 k: np.zeros((1, 47))
81 for k in _COLSNM}
82 reader = csv.reader(io.open(fn, encoding="utf-8-sig"))
83 next(reader)
84 def _f(d):
85 if not d or d == "-":
86 # 不明とか未報告の意味だと思う。つまりこれはゼロではないので、
87 # 可搬性のためには NaN の方が本当はベターだけれど、ここではあまり考えないでおく。
88 return 0
89 return float(d)
90
91 era = datetime.datetime(2020, 1, 16) # NHKまとめのほうの起点日
92 curdate = era
93 for line in reader:
94 date = datetime.datetime(*map(int, line[:3]))
95 dadv = (date - curdate).days
96 data = list(map(_f, line[5:]))
97 p = _PREFCODE[line[3]]
98 for i, k in enumerate(_COLSNM):
99 for _ in range(dadv):
100 result[k] = np.vstack((result[k], np.zeros((1, 47))))
101 if "_movavg" not in k:
102 result[k][-1, p - 1] = data[i]
103 dt = (date - era).days
104 for k in [k for k in _COLSNM if "_movavg" in k]:
105 k1, k2 = k, k.replace("_movavg", "")
106 result[k1][dt, p - 1] = result[k2][
107 max(0, dt - 7):dt + 1, p - 1].mean()
108 curdate = date
109 np.savez(io.open("toyokeizai_online_covid19.npz", "wb"), **result)
110
111
112 if __name__ == '__main__':
113 _tonpa("covid19-master/data/prefectures.csv")
1 # -*- coding: utf-8 -*-
2 import io
3 import datetime
4 import functools
5 import numpy as np
6 from scipy.optimize import curve_fit
7 import matplotlib as mpl
8 import matplotlib.pyplot as plt
9 import matplotlib.font_manager
10
11
12 def _vis(dat, dattko):
13 fontprop = matplotlib.font_manager.FontProperties(
14 fname="c:/Windows/Fonts/meiryo.ttc")
15
16 era = "2020/1/16" # 木曜日
17 era = datetime.datetime(*map(int, era.split("/")))
18 (wh_sl, wh_n) = (slice(12, 12+1), "東京")
19
20 npat = dat["newpat"][:,wh_sl].sum(axis=1)
21 npatma = dat["newpatmovavg"][:,wh_sl].sum(axis=1)
22 #hosp = dattko["hospitalized"][:,wh_sl].sum(axis=1)
23 hospma = dattko["hospitalized_movavg"][:,wh_sl].sum(axis=1)
24 sevma = dattko["serious"][:,wh_sl].sum(axis=1)
25 dif = len(npat) - len(hospma)
26 if dif > 0:
27 # 東洋経済のほうが更新が一日遅い。
28 npat = npat[:-dif]
29 npatma = npatma[:-dif]
30
31 T = [era + datetime.timedelta(t) for t in range(len(npat))]
32 fig, ax1 = plt.subplots(tight_layout=True)
33 fig.set_size_inches(16.53 * 1.2, 11.69)
34 ax2 = ax1.twinx()
35 skip = 7*4*10
36 #
37 # ワイドショーが最近やってる「単純計算でこのまま~%減少ペースならば云々」そのもの
38 # を。簡単のために直近の最大(1月13日)と本日の比較とする。計算は要は「a^t * n0」の
39 # a を求める問題。
40 #
41 n0 = npatma[skip:].max()
42 [t0], = np.where(npatma[skip:] == n0) # 1月13日
43 def _fcurve(r, t, a):
44 return r * np.power(a, t)
45 t1 = len(npatma[skip:]) - 1
46 popt, pcov = curve_fit(
47 functools.partial(_fcurve, (n0,)),
48 np.arange(t1 - t0 + 1), npatma[skip:][t0:])
49 preds = []
50 val = npatma[skip:][t1]
51 while True:
52 preds.append(val)
53 if val < 500.: # 東京の感染者数週平均が500を下回る?
54 break
55 val *= popt[0]
56 #
57 lines = []
58 l, = ax1.plot(T[skip:], npatma[skip:])
59 lines.append(l)
60 l, = ax1.plot(
61 [era + datetime.timedelta(skip + t)
62 for t in range(t1, t1 + len(preds))], preds)
63 lines.append(l)
64 #
65 l, = ax2.semilogy(T[skip:], hospma[skip:], color="tab:green", alpha=0.1)
66 ax2.fill_between(T[skip:], hospma[skip:], alpha=0.05, color="tab:green")
67 lines.append(l)
68 #
69 hn0 = hospma[skip:].max()
70 [hmax], = np.where(hospma[skip:] == hn0)
71 popt2, pcov2 = curve_fit(
72 functools.partial(_fcurve, (hn0,)),
73 np.arange(t1 - hmax + 1), hospma[skip:][hmax:])
74 l, = ax2.plot(
75 [era + datetime.timedelta(skip + t)
76 for t in range(t1, t1 + len(preds))],
77 [_fcurve(hospma[skip:][t1], t - t1, popt2[0])
78 for t in range(t1, t1 + len(preds))],
79 "g", alpha=0.1)
80 lines.append(l)
81 l, = ax2.semilogy(T[skip:], sevma[skip:], color="tab:purple", alpha=0.5)
82 ax2.fill_between(T[skip:], sevma[skip:], alpha=0.05, color="tab:purple")
83 lines.append(l)
84 #
85 ax1.legend(
86 lines,
87 ["新規陽性判明者数(7日移動平均実績)",
88 "減少率{:.2f}(per days)での週平均予測".format(popt[0]),
89 "入院治療等を要する者(7日移動平均実績)",
90 "減少率{:.2f}(per days)での入院治療等を要する者週平均予測".format(popt2[0]),
91 "重傷者数(7日移動平均実績)",
92 ],
93 shadow=True, prop=fontprop)
94 ax1.set_xlabel(
95 """{}の新規感染者数推移
96 (「NHK新型コロナウイルス関連データ」と「東洋経済オンライン「新型コロナウイルス 国内感染の状況」制作:荻原和樹」を加工)""".format(
97 wh_n),
98 fontproperties=fontprop)
99 ax1.set_ylabel("新規陽性判明者数", fontproperties=fontprop)
100 ax2.set_ylabel("入院治療等を要する者", fontproperties=fontprop)
101 ax1.axhspan(0, 500, facecolor="lightgray", alpha=0.1)
102 fig.autofmt_xdate()
103 #
104 tlast = era + datetime.timedelta(skip + t1 + len(preds) - 1)
105 #
106 for evtxt, evdt in (
107 ("GoToトラベル全国一時停止", datetime.datetime(2020, 12, 15)),
108 ("1都3県に緊急事態宣言", datetime.datetime(2021, 1, 7)),
109 ("緊急事態宣言11都府県に拡大", datetime.datetime(2021, 1, 13)),
110 ("東京の週平均が500を下回る?", tlast),
111 ("", T[skip + hmax]),
112 ):
113 ax1.annotate("{} ({})".format(evtxt, str(evdt)[:len("????-??-??")]),
114 xy=(evdt, 0), xycoords='data',
115 xytext=(
116 0.5,
117 (evdt - era).days / (tlast - era).days),
118 textcoords='axes fraction',
119 arrowprops=dict(facecolor='black', width=0.0001, alpha=0.1),
120 horizontalalignment='right', verticalalignment='top',
121 alpha=0.3,
122 fontproperties=fontprop)
123 ax1.vlines([evdt], 0, 1, transform=ax1.get_xaxis_transform(), alpha=0.1)
124 #
125 #plt.show()
126 fig.savefig(
127 "nhk_news_covid19_prefectures_daily_data5_2_5.png")
128 plt.close(fig)
129
130
131 if __name__ == '__main__':
132 fn1 = "nhk_news_covid19_prefectures_daily_data.npz"
133 fn2 = "toyokeizai_online_covid19.npz"
134 _vis(np.load(io.open(fn1, "rb")), np.load(io.open(fn2, "rb")))
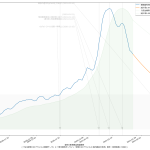
2021-01-31 02:20追記:
NHKまとめデータがさっき更新されたついで:
1 # -*- coding: utf-8 -*-
2 import io
3 import datetime
4 import functools
5 import numpy as np
6 from scipy.optimize import curve_fit
7 import matplotlib as mpl
8 import matplotlib.pyplot as plt
9 import matplotlib.font_manager
10
11
12 def _vis(dat, dattko):
13 fontprop = matplotlib.font_manager.FontProperties(
14 fname="c:/Windows/Fonts/meiryo.ttc")
15
16 era = "2020/1/16" # 木曜日
17 era = datetime.datetime(*map(int, era.split("/")))
18 (wh_sl, wh_n) = (slice(12, 12+1), "東京")
19
20 npat = dat["newpat"][:,wh_sl].sum(axis=1)
21 npatma = dat["newpatmovavg"][:,wh_sl].sum(axis=1)
22 #hosp = dattko["hospitalized"][:,wh_sl].sum(axis=1)
23 hospma = dattko["hospitalized_movavg"][:,wh_sl].sum(axis=1)
24 sevma = dattko["serious"][:,wh_sl].sum(axis=1)
25 tkodif = len(npat) - len(hospma) # 東洋経済のほうが更新が一日遅い。
26 #
27 T = [era + datetime.timedelta(t) for t in range(len(npat))]
28 fig, ax1 = plt.subplots(tight_layout=True)
29 fig.set_size_inches(16.53 * 1.2, 11.69)
30 ax2 = ax1.twinx()
31 skip = 7*4*10
32 #
33 # ワイドショーが最近やってる「単純計算でこのまま~%減少ペースならば云々」そのもの
34 # を。簡単のために直近の最大(1月13日)と本日の比較とする。計算は要は「a^t * n0」の
35 # a を求める問題。
36 #
37 n0 = npatma[skip:].max()
38 [t0], = np.where(npatma[skip:] == n0) # 1月13日
39 def _fcurve(r, t, a):
40 return r * np.power(a, t)
41 t1 = len(npatma[skip:]) - 1
42 popt, pcov = curve_fit(
43 functools.partial(_fcurve, (n0,)),
44 np.arange(t1 - t0 + 1), npatma[skip:][t0:])
45 preds = []
46 val = npatma[skip:][t1]
47 while True:
48 preds.append(val)
49 if val < 500.: # 東京の感染者数週平均が500を下回る?
50 break
51 val *= popt[0]
52 #
53 lines = []
54 l, = ax1.plot(T[skip:], npatma[skip:])
55 lines.append(l)
56 l, = ax1.plot(
57 [era + datetime.timedelta(skip + t)
58 for t in range(t1, t1 + len(preds))], preds)
59 lines.append(l)
60 #
61 l, = ax2.semilogy(T[skip:][:-tkodif], hospma[skip:], color="tab:green", alpha=0.1)
62 ax2.fill_between(T[skip:][:-tkodif], hospma[skip:], alpha=0.05, color="tab:green")
63 lines.append(l)
64 #
65 hn0 = hospma[skip:].max()
66 [hmax], = np.where(hospma[skip:] == hn0)
67 popt2, pcov2 = curve_fit(
68 functools.partial(_fcurve, (hn0,)),
69 np.arange(t1 - hmax + 1)[:-tkodif], hospma[skip:][hmax:])
70 l, = ax2.plot(
71 [era + datetime.timedelta(skip + t)
72 for t in range(t1 - tkodif, t1 + len(preds) - tkodif)],
73 [_fcurve(hospma[skip:][t1 - tkodif], t - t1, popt2[0])
74 for t in range(t1 - tkodif, t1 + len(preds) - tkodif)],
75 "g", alpha=0.1)
76 lines.append(l)
77 l, = ax2.semilogy(T[skip:][:-tkodif], sevma[skip:], color="tab:purple", alpha=0.5)
78 ax2.fill_between(T[skip:][:-tkodif], sevma[skip:], alpha=0.05, color="tab:purple")
79 lines.append(l)
80 #
81 ax1.legend(
82 lines,
83 ["新規陽性判明者数(7日移動平均実績)",
84 "減少率{:.2f}(per days)での週平均予測".format(popt[0]),
85 "入院治療等を要する者(7日移動平均実績)",
86 "減少率{:.2f}(per days)での入院治療等を要する者週平均予測".format(popt2[0]),
87 "重傷者数(7日移動平均実績)",
88 ],
89 shadow=True, prop=fontprop)
90 ax1.set_xlabel(
91 """{}の新規感染者数推移
92 (「NHK新型コロナウイルス関連データ」と「東洋経済オンライン「新型コロナウイルス 国内感染の状況」制作:荻原和樹」を加工)""".format(
93 wh_n),
94 fontproperties=fontprop)
95 ax1.set_ylabel("新規陽性判明者数", fontproperties=fontprop)
96 ax2.set_ylabel("入院治療等を要する者", fontproperties=fontprop)
97 ax1.axhspan(0, 500, facecolor="lightgray", alpha=0.1)
98 fig.autofmt_xdate()
99 #
100 tlast = era + datetime.timedelta(skip + t1 + len(preds) - 1)
101 #
102 for evtxt, evdt in (
103 ("GoToトラベル全国一時停止", datetime.datetime(2020, 12, 15)),
104 ("1都3県に緊急事態宣言", datetime.datetime(2021, 1, 7)),
105 ("緊急事態宣言11都府県に拡大", datetime.datetime(2021, 1, 13)),
106 ("東京の週平均が500を下回る?", tlast),
107 ("", T[skip + hmax]),
108 ("", T[-1]),
109 ):
110 ax1.annotate("{} ({})".format(evtxt, str(evdt)[:len("????-??-??")]),
111 xy=(evdt, 0), xycoords='data',
112 xytext=(
113 0.5,
114 (evdt - era).days / (tlast - era).days),
115 textcoords='axes fraction',
116 arrowprops=dict(facecolor='black', width=0.0001, alpha=0.1),
117 horizontalalignment='right', verticalalignment='top',
118 alpha=0.3,
119 fontproperties=fontprop)
120 ax1.vlines([evdt], 0, 1, transform=ax1.get_xaxis_transform(), alpha=0.1 if evtxt else 0.2)
121 #
122 #plt.show()
123 fig.savefig(
124 "nhk_news_covid19_prefectures_daily_data5_2_6.png")
125 plt.close(fig)
126
127
128 if __name__ == '__main__':
129 fn1 = "nhk_news_covid19_prefectures_daily_data.npz"
130 fn2 = "toyokeizai_online_covid19.npz"
131 _vis(np.load(io.open(fn1, "rb")), np.load(io.open(fn2, "rb")))
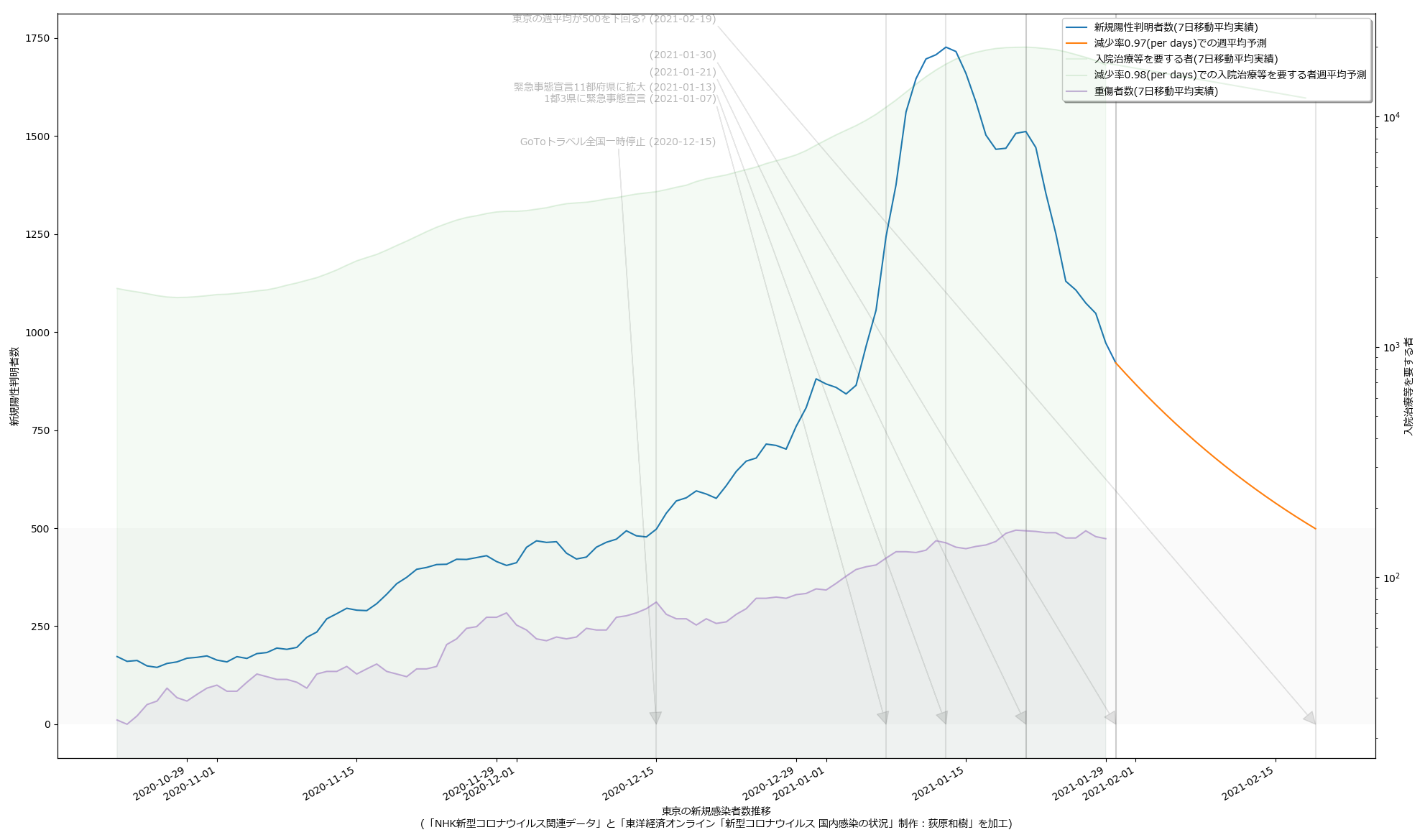
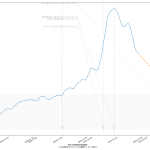
500を下回る予想日が少しずつ早まってる…から喜べ、てのは厳密にはちょっと違う。「n0 * a^t」という関数近似の適用段階で n0 と t0 をスライドさせるかどうかで結果が違う…っていうと通じるかなぁ? 本来は1月13日からの曲線をそのまま延伸するのがより「素直な」予測曲線で、これは今日で言えば1月30日が不連続となる。スクリプトでやってるのは n0 と t0 を 1月30日として計算してる。まぁなんにせよ最初から「信頼するなよ」と言っているインチキな予測ではある。ただ、こうやって日々の予測が前倒ししていってるのは近似した予測曲線よりも下回ったことの現れであることは確かで、なので、正しい意味でなくてもそれそのものは喜んでいい。
2021-02-01 01:10追記:
宣言が3月7日まで延長されそうだ、とのことで:
1 # -*- coding: utf-8 -*-
2 import io
3 import datetime
4 import functools
5 import numpy as np
6 from scipy.optimize import curve_fit
7 import matplotlib as mpl
8 import matplotlib.pyplot as plt
9 import matplotlib.font_manager
10
11
12 def _vis(dat, dattko):
13 fontprop = matplotlib.font_manager.FontProperties(
14 fname="c:/Windows/Fonts/meiryo.ttc")
15
16 era = "2020/1/16" # 木曜日
17 era = datetime.datetime(*map(int, era.split("/")))
18 (wh_sl, wh_n) = (slice(12, 12+1), "東京")
19
20 npat = dat["newpat"][:,wh_sl].sum(axis=1)
21 npatma = dat["newpatmovavg"][:,wh_sl].sum(axis=1)
22 #hosp = dattko["hospitalized"][:,wh_sl].sum(axis=1)
23 hospma = dattko["hospitalized_movavg"][:,wh_sl].sum(axis=1)
24 sevma = dattko["serious"][:,wh_sl].sum(axis=1)
25 tkodif = len(npat) - len(hospma) # 東洋経済のほうが更新が一日遅い。
26 #
27 T = [era + datetime.timedelta(t) for t in range(len(npat))]
28 fig, ax1 = plt.subplots(tight_layout=True)
29 fig.set_size_inches(16.53 * 1.2, 11.69)
30 ax2 = ax1.twinx()
31 skip = 7*4*10 + 14
32 #
33 # ワイドショーが最近やってる「単純計算でこのまま~%減少ペースならば云々」そのもの
34 # を。簡単のために直近の最大(1月13日)と本日の比較とする。計算は要は「a^t * n0」の
35 # a を求める問題。
36 #
37 n0 = npatma[skip:].max()
38 [t0], = np.where(npatma[skip:] == n0) # 1月13日
39 def _fcurve(r, t, a):
40 return r * np.power(a, t)
41 t1 = len(npatma[skip:]) - 1
42 popt, pcov = curve_fit(
43 functools.partial(_fcurve, (n0,)),
44 np.arange(t1 - t0 + 1), npatma[skip:][t0:])
45 daysto500 = 0
46 val = npatma[skip:][t1]
47 while True:
48 if val < 500.: # 東京の感染者数週平均が500を下回る?
49 break
50 val *= popt[0]
51 daysto500 += 1
52 #
53 lines = []
54 l, = ax1.plot(T[skip:], npatma[skip:])
55 lines.append(l)
56 #trng = range(t1, t1 + daysto500)
57 trng = range(t1, (datetime.datetime(2021, 3, 7) - era).days - skip)
58 #
59 l, = ax1.plot(
60 [era + datetime.timedelta(skip + t) for t in trng],
61 [_fcurve(npatma[skip:][t1], t - t1, popt[0]) for t in trng])
62 lines.append(l)
63 #
64 l, = ax2.semilogy(T[skip:][:-tkodif], hospma[skip:], color="tab:green", alpha=0.1)
65 ax2.fill_between(T[skip:][:-tkodif], hospma[skip:], alpha=0.05, color="tab:green")
66 lines.append(l)
67 #
68 hn0 = hospma[skip:].max()
69 [hmax], = np.where(hospma[skip:] == hn0)
70 popt2, pcov2 = curve_fit(
71 functools.partial(_fcurve, (hn0,)),
72 np.arange(t1 - hmax + 1)[:-tkodif], hospma[skip:][hmax:])
73 #trng = range(t1 - tkodif, t1 + daysto500 - tkodif)
74 trng = range(t1 - tkodif, ((datetime.datetime(2021, 3, 7) - era).days - skip) - tkodif)
75 l, = ax2.plot(
76 [era + datetime.timedelta(skip + t) for t in trng],
77 [_fcurve(hospma[skip:][t1 - tkodif], t - (t1 - tkodif), popt2[0]) for t in trng],
78 "g", alpha=0.1)
79 lines.append(l)
80 l, = ax2.semilogy(T[skip:][:-tkodif], sevma[skip:], color="tab:purple", alpha=0.5)
81 ax2.fill_between(T[skip:][:-tkodif], sevma[skip:], alpha=0.05, color="tab:purple")
82 lines.append(l)
83 #
84 ax1.legend(
85 lines,
86 ["新規陽性判明者数(7日移動平均実績)",
87 "減少率{:.2f}(per days)での週平均予測".format(popt[0]),
88 "入院治療等を要する者(7日移動平均実績)",
89 "減少率{:.2f}(per days)での入院治療等を要する者週平均予測".format(popt2[0]),
90 "重傷者数(7日移動平均実績)",
91 ],
92 shadow=True, prop=fontprop)
93 ax1.set_xlabel(
94 """{}の新規感染者数推移
95 (「NHK新型コロナウイルス関連データ」と「東洋経済オンライン「新型コロナウイルス 国内感染の状況」制作:荻原和樹」を加工)""".format(
96 wh_n),
97 fontproperties=fontprop)
98 ax1.set_ylabel("新規陽性判明者数", fontproperties=fontprop)
99 ax2.set_ylabel("入院治療等を要する者", fontproperties=fontprop)
100 ax1.axhspan(0, 500, facecolor="lightgray", alpha=0.1)
101 fig.autofmt_xdate()
102 #
103 tlast = era + datetime.timedelta(skip + t1 + daysto500 - 1)
104 #
105 for evtxt, evdt in (
106 ("GoToトラベル全国一時停止", datetime.datetime(2020, 12, 15)),
107 ("1都3県に緊急事態宣言", datetime.datetime(2021, 1, 7)),
108 ("緊急事態宣言11都府県に拡大", datetime.datetime(2021, 1, 13)),
109 ("東京の週平均が500を下回る?", tlast),
110 ("", T[skip + hmax]),
111 ("", T[-1]),
112 ("緊急事態宣言当初期限", datetime.datetime(2021, 2, 7)),
113 ("緊急事態宣言再設定後期限?", datetime.datetime(2021, 3, 7)),
114 ):
115 ax1.annotate("{} ({})".format(evtxt, str(evdt)[:len("????-??-??")]),
116 xy=(evdt, 0), xycoords='data',
117 xytext=(
118 0.4,
119 ((evdt - era).days / (tlast - era).days) - 0.7),
120 textcoords='axes fraction',
121 arrowprops=dict(facecolor='black', width=0.0001, alpha=0.1),
122 horizontalalignment='right', verticalalignment='top',
123 alpha=0.3,
124 fontproperties=fontprop)
125 ax1.vlines([evdt], 0, 1, transform=ax1.get_xaxis_transform(), alpha=0.1 if evtxt else 0.2)
126 #
127 #plt.show()
128 fig.savefig(
129 "nhk_news_covid19_prefectures_daily_data5_2_7.png")
130 plt.close(fig)
131
132
133 if __name__ == '__main__':
134 fn1 = "nhk_news_covid19_prefectures_daily_data.npz"
135 fn2 = "toyokeizai_online_covid19.npz"
136 _vis(np.load(io.open(fn1, "rb")), np.load(io.open(fn2, "rb")))
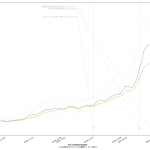
しょうもないミスもあったので直してたりもするが、本質的でもないので説明は割愛。最新データでこんな:
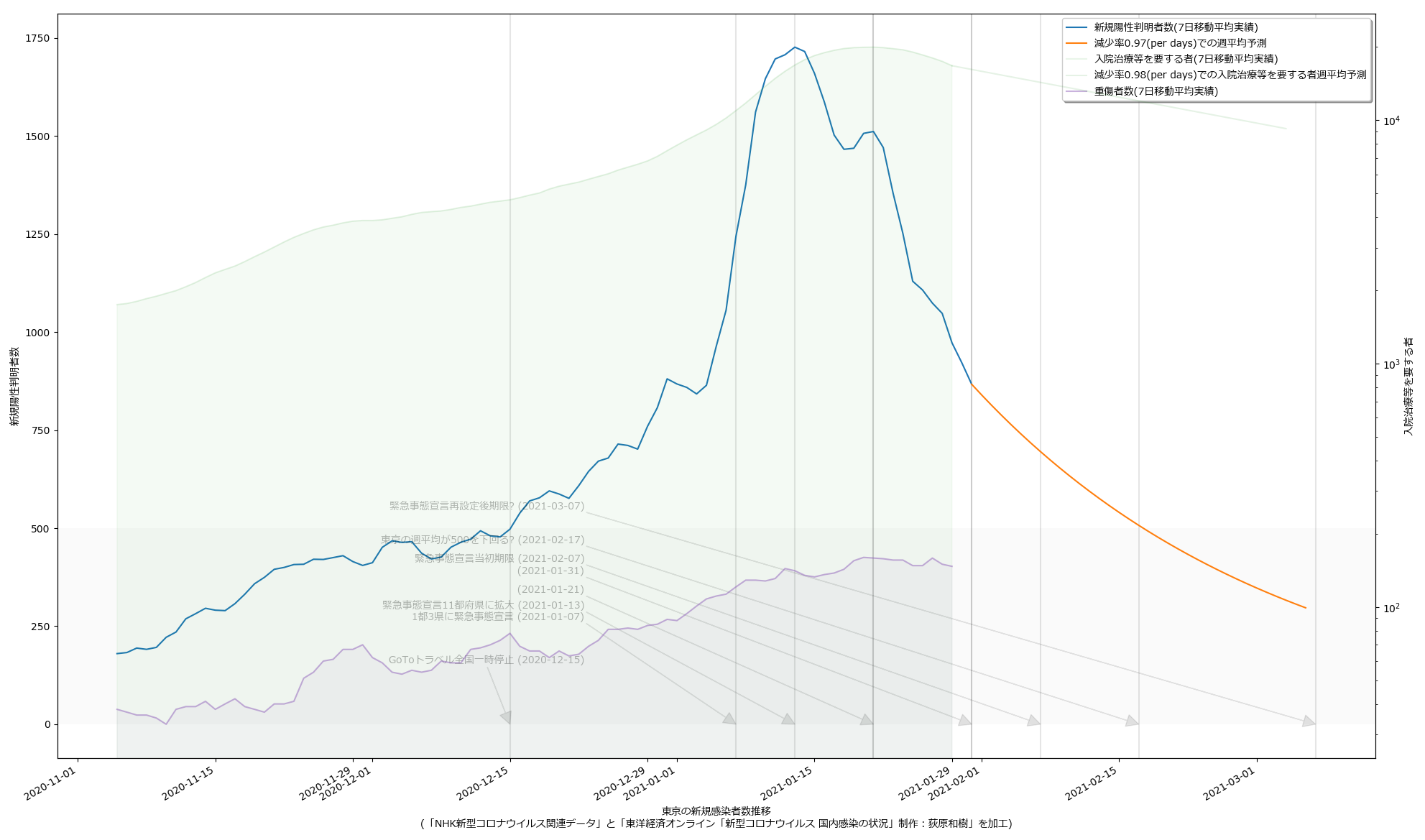
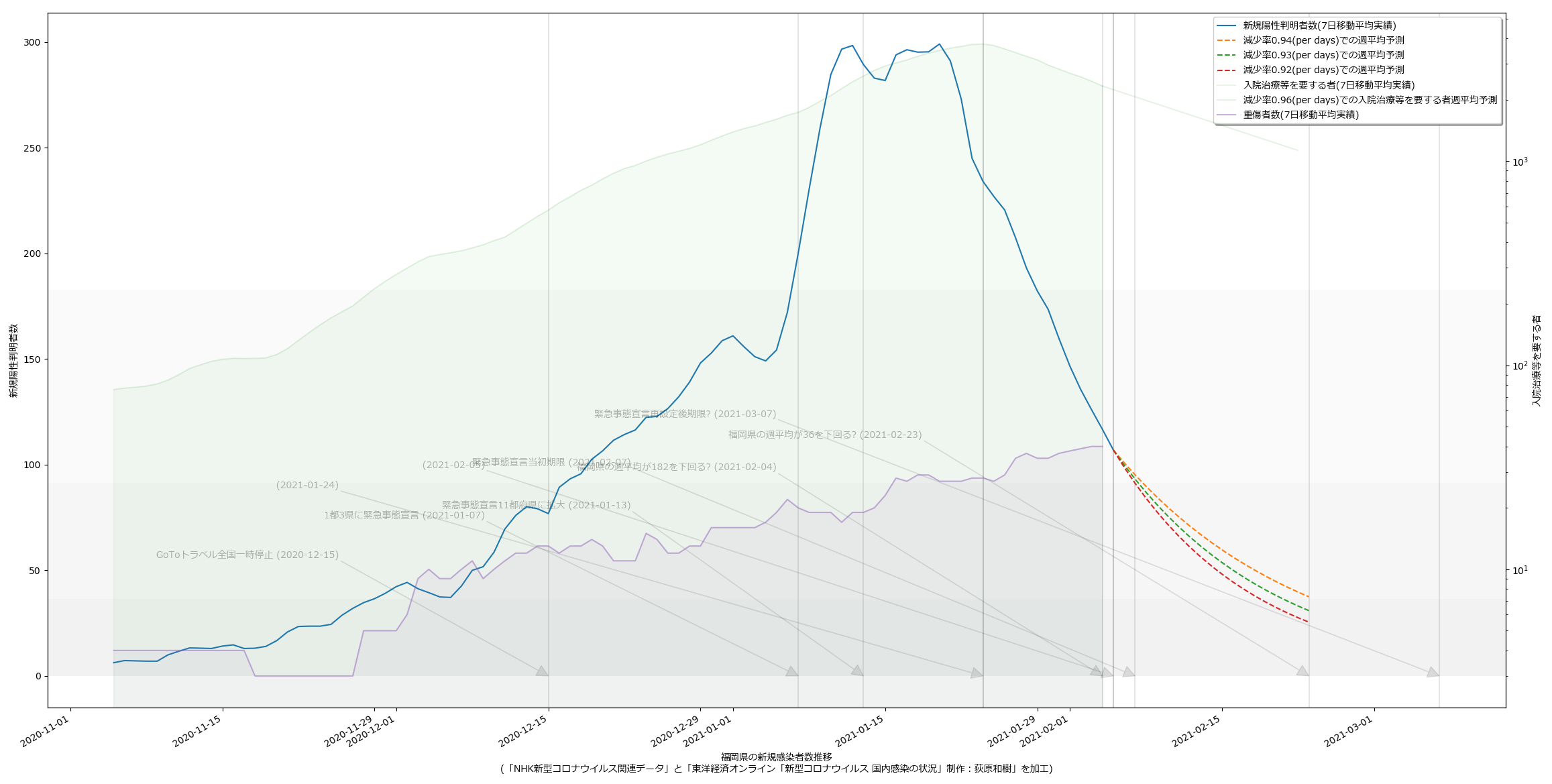
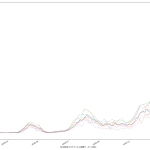
ワタシの予測は「アホ」なので、一切信頼しないで欲しい、のだけれど、まかり間違ってこの予測が遠からないんだとすると、3月7日でも新規感染者数が250人にしかならず、「入院治療等を要する者」も9000人近くいることになり、だとしたらそれでいいのか、ってことにはなる。まぁ日々様子をみてくしかないね。
2021-02-01 09:00追記:
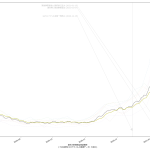
1日100件を下回る水準を目指すべきだ、というのが大曲さんの主張のようで、アホ予測の延伸をしてみる:
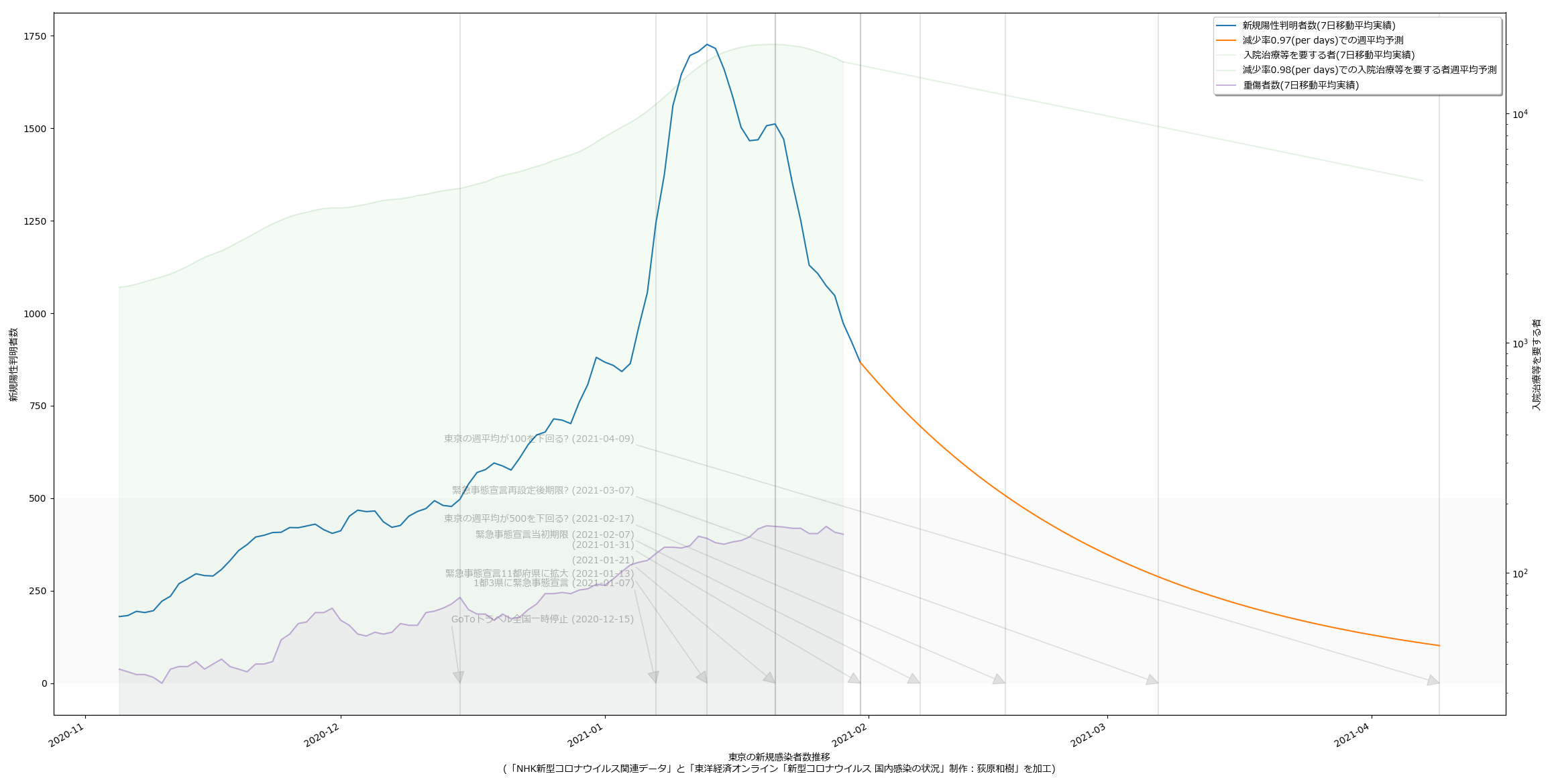
4月9日って…。このアホ予測を後日「やっぱアホだった」とコキおろしたいわけよ、素人予測なんだから当たるわけなかったじゃんか、とね。むろんアホ予測よりもはるかに早く100未満に到達して驚きたい。そうなるといいよな。
にしても、「入院治療等を要する者」が4月9日にも5000人いる、のだとしたら…。考えたくないよなぁ…。
2021-02-02 02:00追記:
「8日平均を計算しちゃってた」問題もあったので、最新データとともに、改めて変更したスクリプトを:
1 # -*- coding: utf-8 -*-
2 import io
3 import sys
4 import csv
5 import datetime
6 import numpy as np
7
8
9 def _tonpa(fn):
10 # rownum=2020/1/16からの日数, colnum=都道府県コード-1
11 result = {
12 "newpat": np.zeros((1, 47)),
13 "newpatmovavg": np.zeros((1, 47)), # 週平均
14 "newpatmovavg2": np.zeros((1, 47)), # 2週平均
15 "newdeath": np.zeros((1, 47)),
16 "newdeathmovavg": np.zeros((1, 47)),
17 "newdeathmovavg2": np.zeros((1, 47)),
18 "effrepro": np.zeros((1, 47)), # 実効再生産数
19 }
20 era = "2020/1/16"
21 era = datetime.datetime(*map(int, era.split("/")))
22 reader = csv.reader(io.open(fn, encoding="utf-8-sig"))
23 next(reader)
24 for row in reader:
25 dt, (p, c, d) = row[0], map(int, row[1::2])
26 dt = (datetime.datetime(*map(int, dt.split("/"))) - era).days
27 if dt > len(result["newpat"]) - 1:
28 for k in result.keys():
29 result[k] = np.vstack((result[k], np.zeros((1, 47))))
30 result["newpat"][dt, p - 1] = c
31 result["newpatmovavg"][dt, p - 1] = result[
32 "newpat"][max(0, dt-6):dt+1, p - 1].mean()
33 result["newpatmovavg2"][dt, p - 1] = result[
34 "newpat"][max(0, dt-13):dt+1, p - 1].mean()
35 result["newdeath"][dt, p - 1] = d
36 result["newdeathmovavg"][dt, p - 1] = result[
37 "newdeath"][max(0, dt-6):dt+1, p - 1].mean()
38 result["newdeathmovavg2"][dt, p - 1] = result[
39 "newdeath"][max(0, dt-13):dt+1, p - 1].mean()
40 # 実効再生産数は、現京都大学西浦教授提案による簡易計算(を一般向け説明にまとめてくれ
41 # ている東洋経済ONLINE)より:
42 # (直近7日間の新規陽性者数/その前7日間の新規陽性者数)^(平均世代時間/報告間隔)
43 # 平均世代時間は=5日、報告間隔=7日の仮定。
44 # のつもりだが、東洋経済ONLINEで GitHub にて公開(閉鎖予定)されてる計算結果の csv とは
45 # 値が違う。違うことそのものは致し方ないこと。計算タイミングによって結果は変わるし、
46 # NHK まとめと東洋経済ONLINEのデータはシンクロしてないから。思いっきり間違ってるって
47 # ことはなさそう。近い値にはなってる。
48 for dt in range(len(result["newpat"])):
49 # 実効再生産数(都道府県別)
50 sum1 = result["newpat"][max(0, dt-6):dt+1, :].sum(axis=0)
51 sum2 = result["newpat"][max(0, dt-6-7):max(0, dt-6)+1, :].sum(axis=0)
52 result["effrepro"][dt, :] = np.power(sum1 / sum2, 5. / 7.)
53 np.savez(io.open("nhk_news_covid19_prefectures_daily_data.npz", "wb"), **result)
54
55
56 if __name__ == '__main__':
57 fn = "nhk_news_covid19_prefectures_daily_data.csv"
58 _tonpa(fn)
1 # -*- coding: utf-8-unix -*-
2 import io
3 import csv
4 import datetime
5 import numpy as np
6
7
8 _PREFCODE = {
9 '北海道': 1,
10 '青森県': 2,
11 '岩手県': 3,
12 '宮城県': 4,
13 '秋田県': 5,
14 '山形県': 6,
15 '福島県': 7,
16 '茨城県': 8,
17 '栃木県': 9,
18 '群馬県': 10,
19 '埼玉県': 11,
20 '千葉県': 12,
21 '東京都': 13,
22 '神奈川県': 14,
23 '新潟県': 15,
24 '富山県': 16,
25 '石川県': 17,
26 '福井県': 18,
27 '山梨県': 19,
28 '長野県': 20,
29 '岐阜県': 21,
30 '静岡県': 22,
31 '愛知県': 23,
32 '三重県': 24,
33 '滋賀県': 25,
34 '京都府': 26,
35 '大阪府': 27,
36 '兵庫県': 28,
37 '奈良県': 29,
38 '和歌山県': 30,
39 '鳥取県': 31,
40 '島根県': 32,
41 '岡山県': 33,
42 '広島県': 34,
43 '山口県': 35,
44 '徳島県': 36,
45 '香川県': 37,
46 '愛媛県': 38,
47 '高知県': 39,
48 '福岡県': 40,
49 '佐賀県': 41,
50 '長崎県': 42,
51 '熊本県': 43,
52 '大分県': 44,
53 '宮崎県': 45,
54 '鹿児島県': 46,
55 '沖縄県': 47,
56 }
57 _COLSNM = (
58 "testedPositive",
59 "peopleTested",
60 "hospitalized",
61 "serious",
62 "discharged",
63 "deaths",
64 "effectiveReproductionNumber",
65
66 "hospitalized_movavg",
67 "serious_movavg",
68 )
69 def _tonpa(fn):
70 # 元データ prefectures.csv の著作権:
71 # 『東洋経済オンライン「新型コロナウイルス 国内感染の状況」制作:荻原和樹』
72 #
73 # 1. 開始日が NHK まとめと違う
74 # 2. 更新のない都道府県は省略してある
75 # 3. 日付は一応抜けなく連続している、今のところ
76 #
77 # NHKまとめのほうで密行列として扱った同じノリにしたい、ここでは面倒だけど。
78 # つまり、列方向に都道府県、行方向に日付。
79 result = {
80 k: np.zeros((1, 47))
81 for k in _COLSNM}
82 reader = csv.reader(io.open(fn, encoding="utf-8-sig"))
83 next(reader)
84 def _f(d):
85 if not d or d == "-":
86 # 不明とか未報告の意味だと思う。つまりこれはゼロではないので、
87 # 可搬性のためには NaN の方が本当はベターだけれど、ここではあまり考えないでおく。
88 return 0
89 return float(d)
90
91 era = datetime.datetime(2020, 1, 16) # NHKまとめのほうの起点日
92 curdate = era
93 for line in reader:
94 date = datetime.datetime(*map(int, line[:3]))
95 dadv = (date - curdate).days
96 data = list(map(_f, line[5:]))
97 p = _PREFCODE[line[3]]
98 for i, k in enumerate(_COLSNM):
99 for _ in range(dadv):
100 result[k] = np.vstack((result[k], np.zeros((1, 47))))
101 if "_movavg" not in k:
102 result[k][-1, p - 1] = data[i]
103 dt = (date - era).days
104 for k in [k for k in _COLSNM if "_movavg" in k]:
105 k1, k2 = k, k.replace("_movavg", "")
106 result[k1][dt, p - 1] = result[k2][
107 max(0, dt - 6):dt + 1, p - 1].mean()
108 curdate = date
109 np.savez(io.open("toyokeizai_online_covid19.npz", "wb"), **result)
110
111
112 if __name__ == '__main__':
113 # GitHub 更新は止まったが、https://toyokeizai.net/sp/visual/tko/covid19/ 末尾から
114 # 直接ダウンロードできるようになった。
115 _tonpa("prefectures.csv")
1 # -*- coding: utf-8 -*-
2 import io
3 import datetime
4 import functools
5 import numpy as np
6 from scipy.optimize import curve_fit
7 import matplotlib as mpl
8 import matplotlib.pyplot as plt
9 import matplotlib.font_manager
10
11
12 def _vis(dat, dattko):
13 fontprop = matplotlib.font_manager.FontProperties(
14 fname="c:/Windows/Fonts/meiryo.ttc")
15
16 era = "2020/1/16" # 木曜日
17 era = datetime.datetime(*map(int, era.split("/")))
18 (wh_sl, wh_n) = (slice(12, 12+1), "東京")
19
20 npat = dat["newpat"][:,wh_sl].sum(axis=1)
21 npatma = dat["newpatmovavg"][:,wh_sl].sum(axis=1)
22 #hosp = dattko["hospitalized"][:,wh_sl].sum(axis=1)
23 hospma = dattko["hospitalized_movavg"][:,wh_sl].sum(axis=1)
24 sevma = dattko["serious"][:,wh_sl].sum(axis=1)
25 tkodif = len(npat) - len(hospma) # 東洋経済のほうが更新が一日遅い。
26 #
27 T = [era + datetime.timedelta(t) for t in range(len(npat))]
28 fig, ax1 = plt.subplots(tight_layout=True)
29 fig.set_size_inches(16.53 * 1.4, 11.69)
30 ax2 = ax1.twinx()
31 skip = 7*4*10 + 14
32 #
33 # ワイドショーが最近やってる「単純計算でこのまま~%減少ペースならば云々」そのもの
34 # を。簡単のために直近の最大(1月13日)と本日の比較とする。計算は要は「a^t * n0」の
35 # a を求める問題。
36 #
37 n0 = npatma[skip:].max()
38 [t0], = np.where(npatma[skip:] == n0) # 1月13日
39 def _fcurve(r, t, a):
40 return r * np.power(a, t)
41 t1 = len(npatma[skip:]) - 1
42 popt, pcov = curve_fit(
43 functools.partial(_fcurve, (n0,)),
44 np.arange(t1 - t0 + 1), npatma[skip:][t0:])
45 daysto500 = 0
46 val = npatma[skip:][t1]
47 while True:
48 if val > 500.: # 東京の感染者数週平均が500を下回る?
49 break
50 val *= popt[0]
51 daysto500 += 1
52 daysto100 = daysto500
53 while True:
54 if val > 100.: # 東京の感染者数週平均が100を下回る?
55 break
56 val *= popt[0]
57 daysto100 += 1
58 #
59 lines = []
60 l, = ax1.plot(T[skip:], npatma[skip:])
61 lines.append(l)
62 #trng = range(t1, t1 + daysto500)
63 #trng = range(t1, (datetime.datetime(2021, 3, 7) - era).days - skip)
64 trng = range(t1, t1 + daysto100)
65 #
66 l, = ax1.plot(
67 [era + datetime.timedelta(skip + t) for t in trng],
68 [_fcurve(npatma[skip:][t1], t - t1, popt[0]) for t in trng])
69 lines.append(l)
70 #
71 l, = ax2.semilogy(T[skip:][:-tkodif], hospma[skip:], color="tab:green", alpha=0.1)
72 ax2.fill_between(T[skip:][:-tkodif], hospma[skip:], alpha=0.05, color="tab:green")
73 lines.append(l)
74 #
75 hn0 = hospma[skip:].max()
76 [hmax], = np.where(hospma[skip:] == hn0)
77 popt2, pcov2 = curve_fit(
78 functools.partial(_fcurve, (hn0,)),
79 np.arange(t1 - hmax + 1)[:-tkodif], hospma[skip:][hmax:])
80 #trng = range(t1 - tkodif, t1 + daysto500 - tkodif)
81 #trng = range(t1 - tkodif, ((datetime.datetime(2021, 3, 7) - era).days - skip) - tkodif)
82 trng = range(t1 - tkodif, t1 + daysto100 - tkodif)
83 l, = ax2.plot(
84 [era + datetime.timedelta(skip + t) for t in trng],
85 [_fcurve(hospma[skip:][t1 - tkodif], t - (t1 - tkodif), popt2[0]) for t in trng],
86 "g", alpha=0.1)
87 lines.append(l)
88 l, = ax2.semilogy(T[skip:][:-tkodif], sevma[skip:], color="tab:purple", alpha=0.5)
89 ax2.fill_between(T[skip:][:-tkodif], sevma[skip:], alpha=0.05, color="tab:purple")
90 lines.append(l)
91 #
92 ax1.legend(
93 lines,
94 ["新規陽性判明者数(7日移動平均実績)",
95 "減少率{:.2f}(per days)での週平均予測".format(popt[0]),
96 "入院治療等を要する者(7日移動平均実績)",
97 "減少率{:.2f}(per days)での入院治療等を要する者週平均予測".format(popt2[0]),
98 "重傷者数(7日移動平均実績)",
99 ],
100 shadow=True, prop=fontprop)
101 ax1.set_xlabel(
102 """{}の新規感染者数推移
103 (「NHK新型コロナウイルス関連データ」と「東洋経済オンライン「新型コロナウイルス 国内感染の状況」制作:荻原和樹」を加工)""".format(
104 wh_n),
105 fontproperties=fontprop)
106 ax1.set_ylabel("新規陽性判明者数", fontproperties=fontprop)
107 ax2.set_ylabel("入院治療等を要する者", fontproperties=fontprop)
108 ax1.axhspan(0, 500, facecolor="lightgray", alpha=0.1)
109 fig.autofmt_xdate()
110 #
111 tlast1 = era + datetime.timedelta(skip + t1 + daysto500 - 1)
112 tlast2 = era + datetime.timedelta(skip + t1 + daysto100 - 1)
113 #
114 for evtxt, evdt in (
115 ("GoToトラベル全国一時停止", datetime.datetime(2020, 12, 15)),
116 ("1都3県に緊急事態宣言", datetime.datetime(2021, 1, 7)),
117 ("緊急事態宣言11都府県に拡大", datetime.datetime(2021, 1, 13)),
118 ("東京の週平均が500を下回る?", tlast1),
119 ("東京の週平均が100を下回る?", tlast2),
120 ("", T[skip + hmax]),
121 ("", T[-1]),
122 ("緊急事態宣言当初期限", datetime.datetime(2021, 2, 7)),
123 ("緊急事態宣言再設定後期限?", datetime.datetime(2021, 3, 7)),
124 ):
125 ax1.annotate("{} ({})".format(evtxt, str(evdt)[:len("????-??-??")]),
126 xy=(evdt, 0), xycoords='data',
127 xytext=(
128 0.4,
129 ((evdt - era).days / (tlast2 - era).days) - 0.6),
130 textcoords='axes fraction',
131 arrowprops=dict(facecolor='black', width=0.0001, alpha=0.1),
132 horizontalalignment='right', verticalalignment='top',
133 alpha=0.3,
134 fontproperties=fontprop)
135 ax1.vlines([evdt], 0, 1, transform=ax1.get_xaxis_transform(), alpha=0.1 if evtxt else 0.2)
136 #
137 #plt.show()
138 fig.savefig(
139 "nhk_news_covid19_prefectures_daily_data5_2_8.png")
140 plt.close(fig)
141
142
143 if __name__ == '__main__':
144 fn1 = "nhk_news_covid19_prefectures_daily_data.npz"
145 fn2 = "toyokeizai_online_covid19.npz"
146 _vis(np.load(io.open(fn1, "rb")), np.load(io.open(fn2, "rb")))
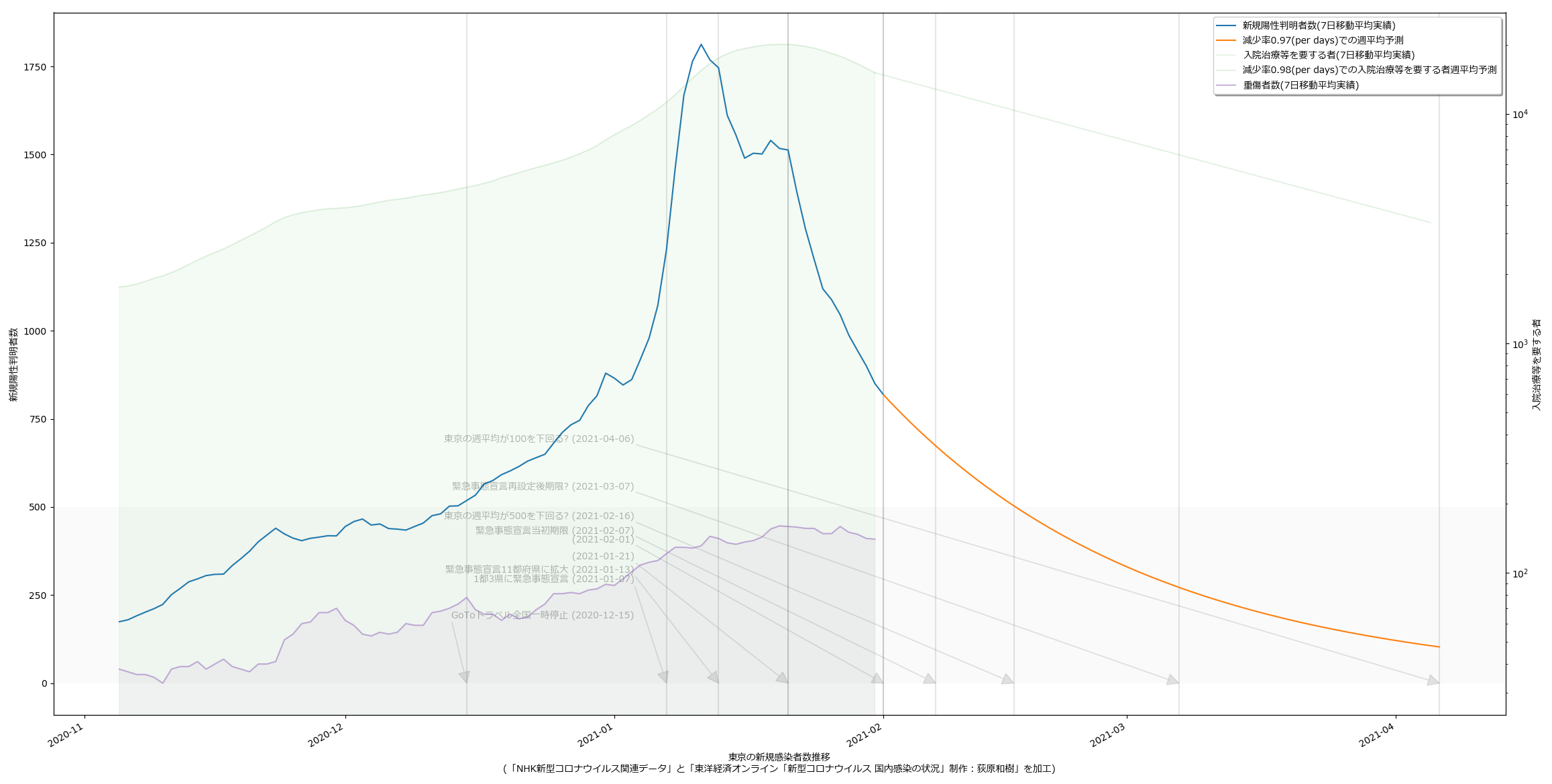
改めて言っておくけれど、「なんで報道(特にテレビ)は、宣言解除の出口のための予測しか伝えようとしないのか」ということね。ここ数日、「いつ宣言を解除すべきか」の議論のために専門家の予測をようやく紹介するようになったが、これって第一波のときと全く同じ流れになってて、成長してないよな一年間何やってたんだ、と思うわけだよ。なんで「このままいくとヤバい」という注意喚起のための予測を報道しないのだ。ほんとよくわかんない。
そしてこれも改めて。「二週間前の行動が今日の新規陽性者数に反映される」というのはつまり端的にいえばこういう意味なのだよ:
- 本日の値をみて驚く→二周回遅れ
- 来週の値を予測して驚く→周回遅れ
- 再来週の値を予測して驚く→やっと周回
ゆえに、なんとなく「未来予測に基づいて行動する=先回り」とみなされそうな気がするけど実際はそうではなくて、本来は「二週間後予測に基づいて行動決定をする」のがようやく最低限で、「三週間後以降予測に基づいて行動を決める」のがやっとこ「先回り」なのだ、てこと、そして「一週間後予測に基づいて行動決定をする」のは本当はこれでも「後手」ではあるんだけれど、そうしないよりは何万倍かはマシで、そして「政権はそれをしてる気配がなく、だから周回遅れどころか二周回遅れになっていて、しかもその自覚がない」ということを問題視していて、そして、もとをただせば「報道が伝えようとしないからだろ?」てこと。
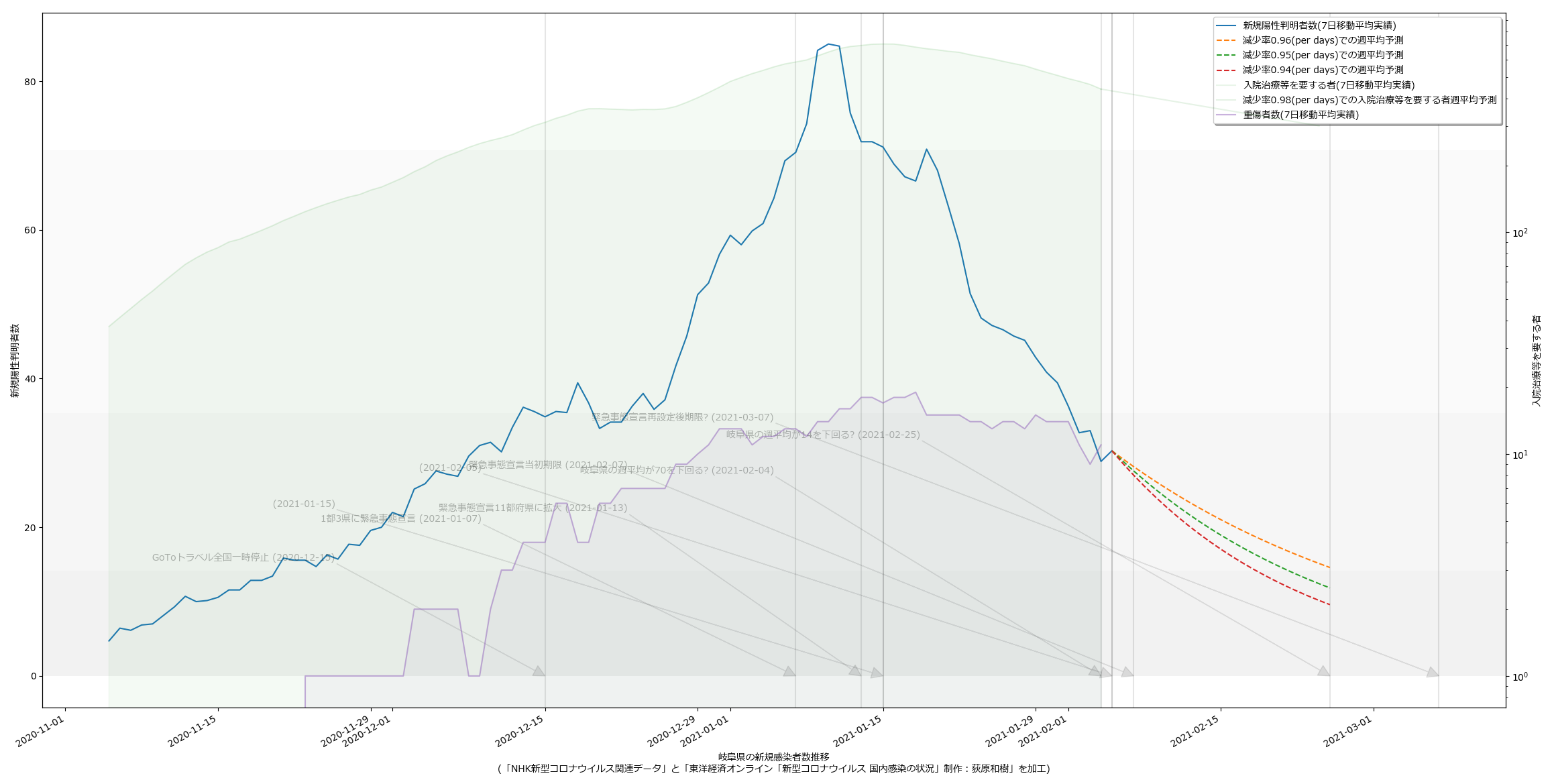
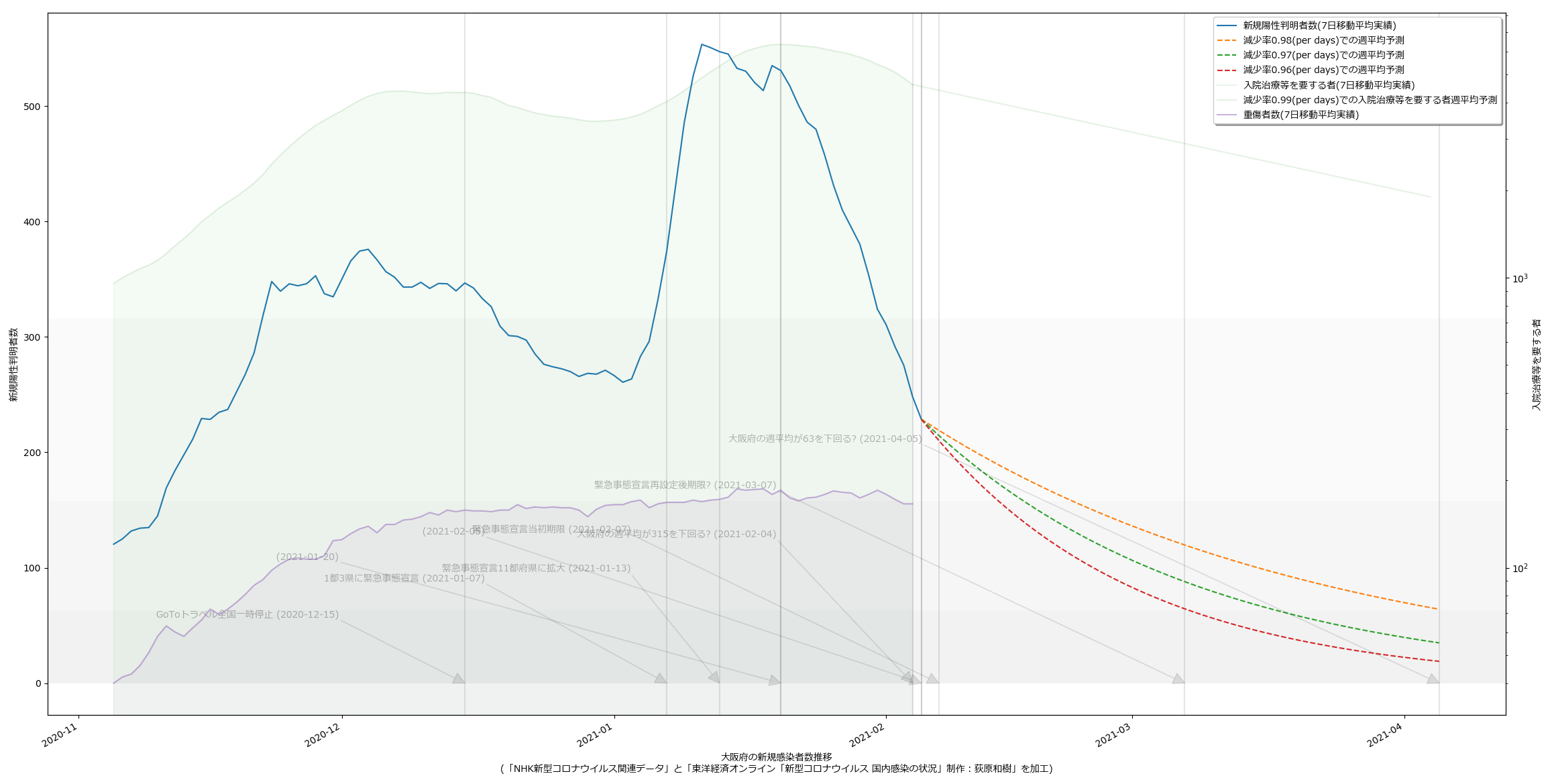
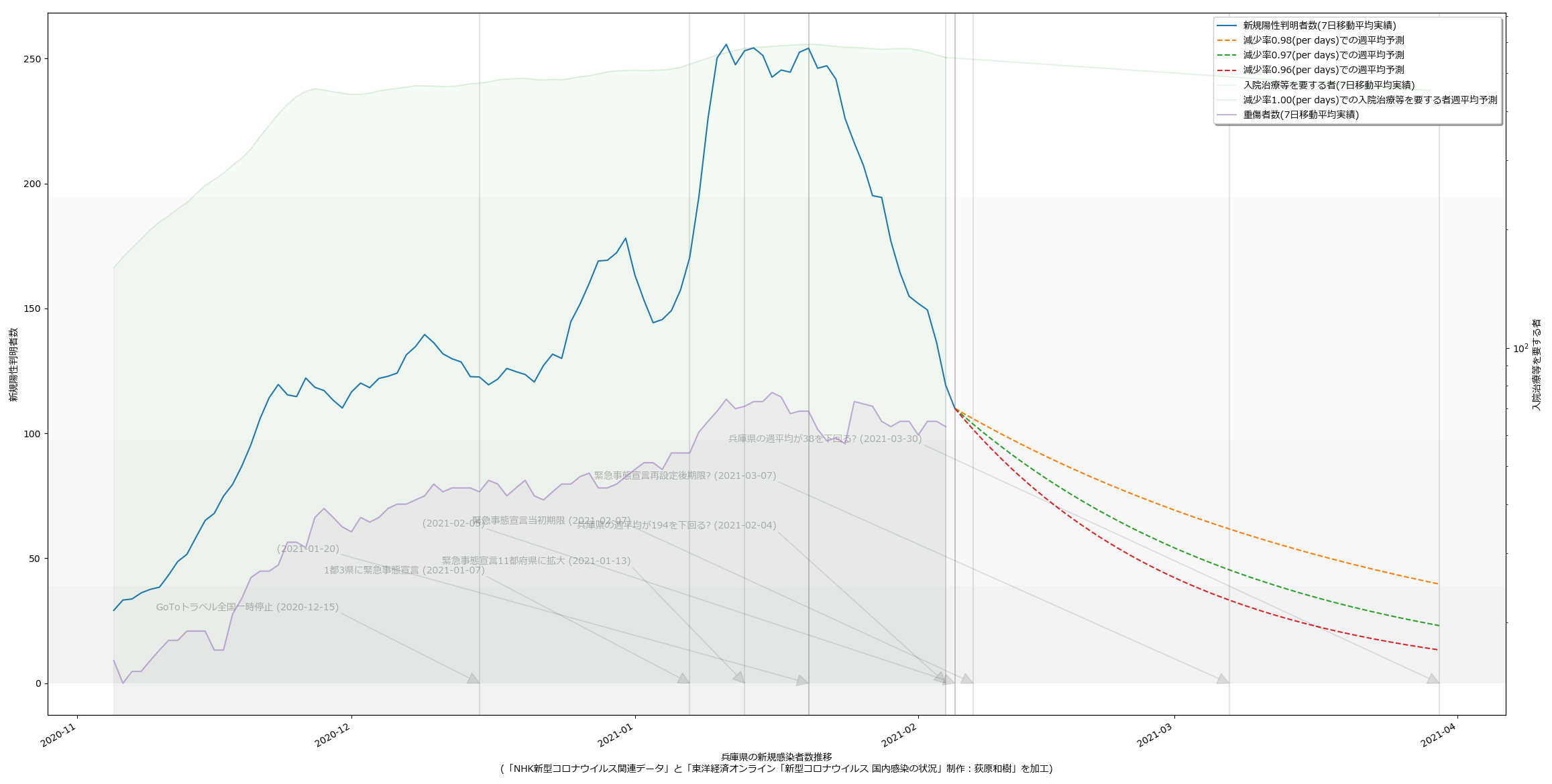
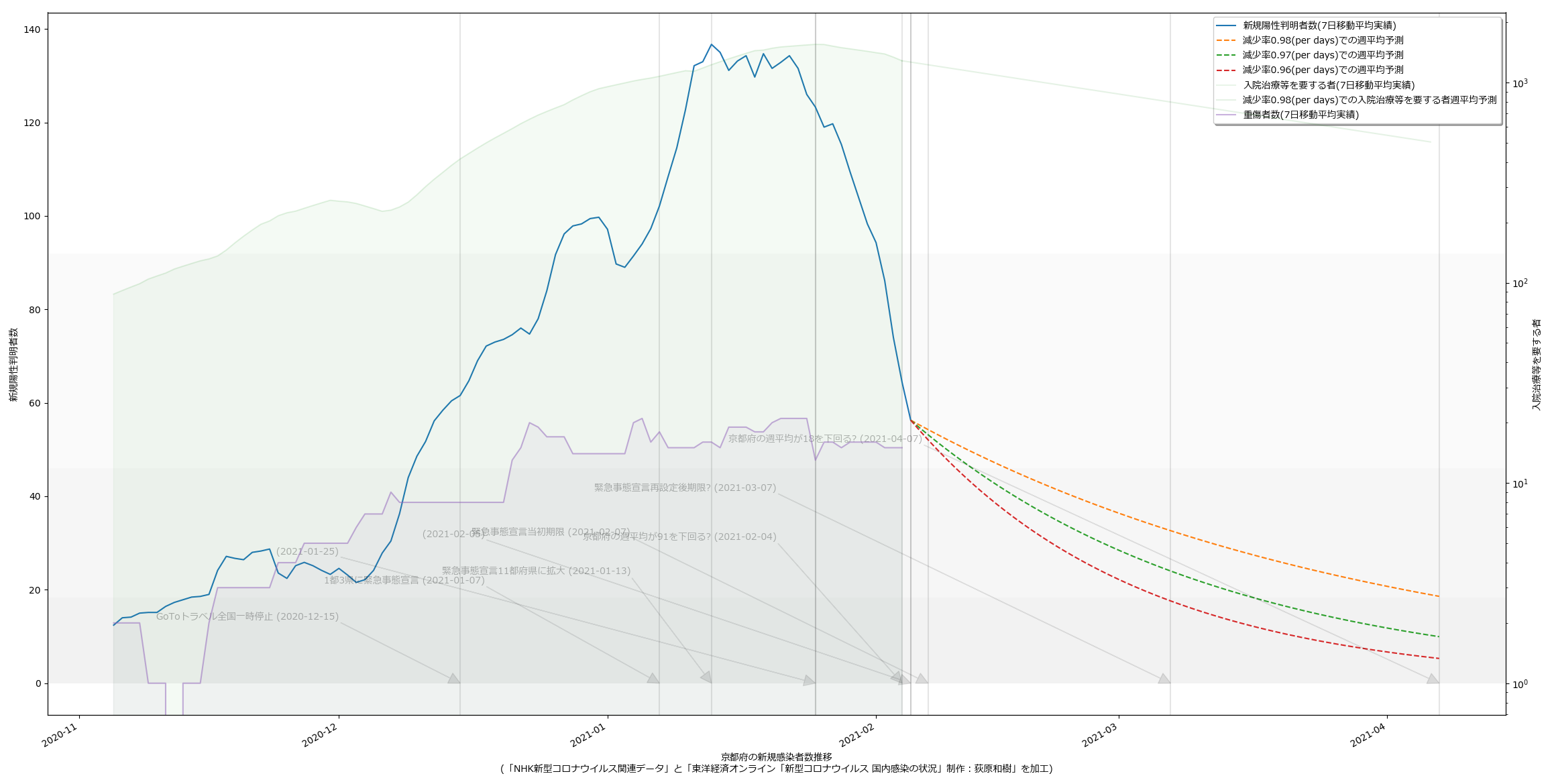
2021-02-06追記:
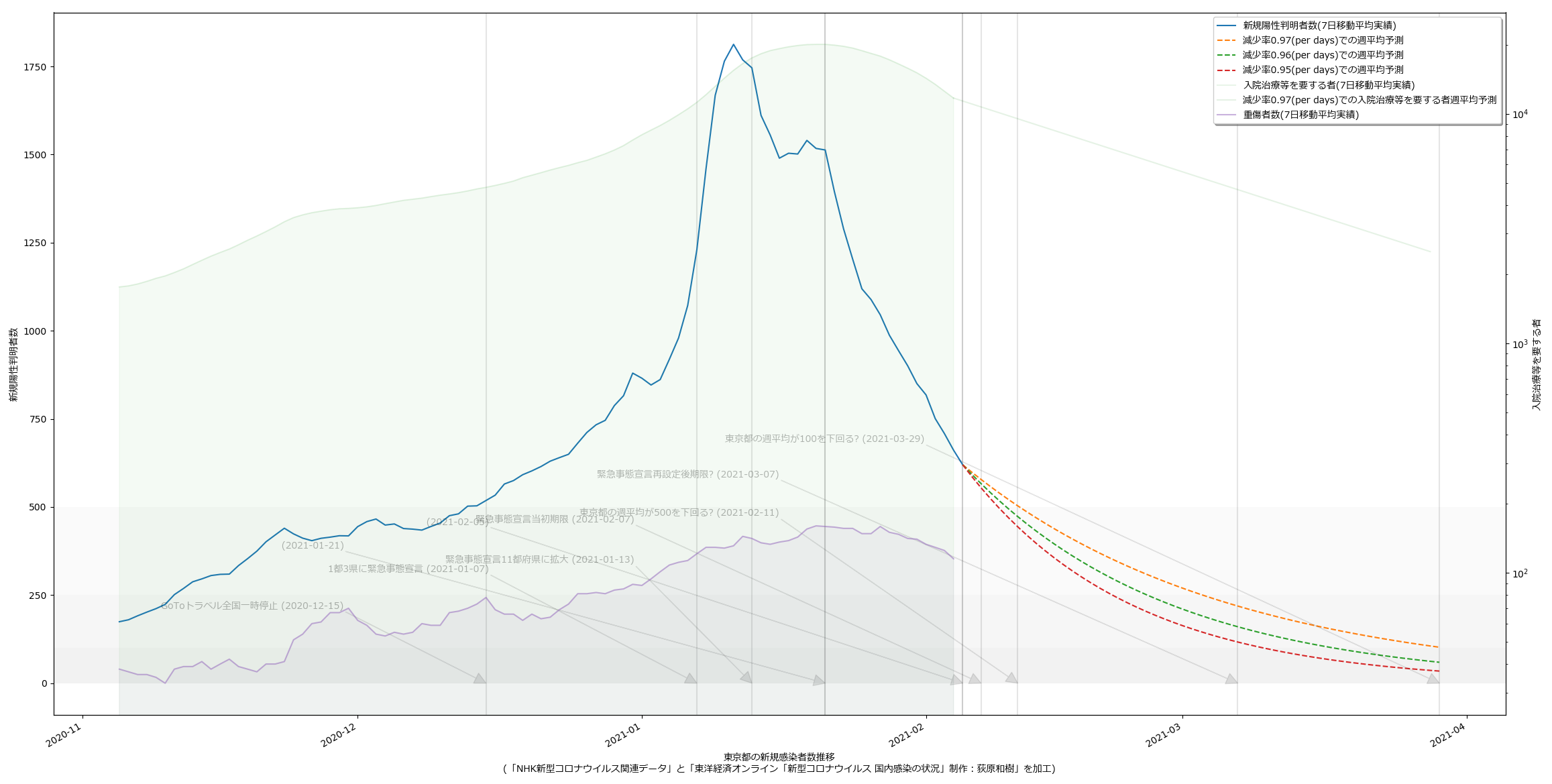
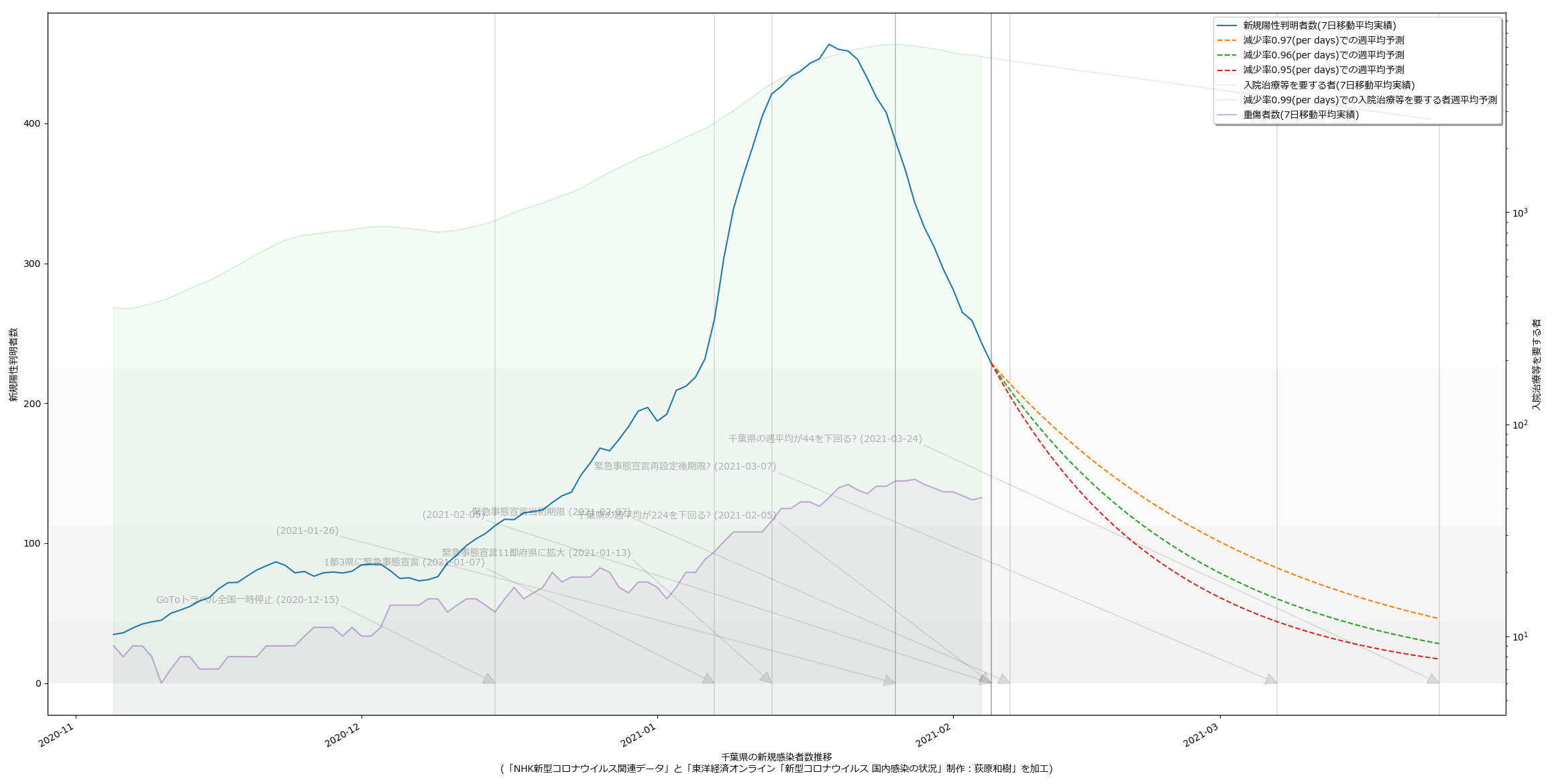
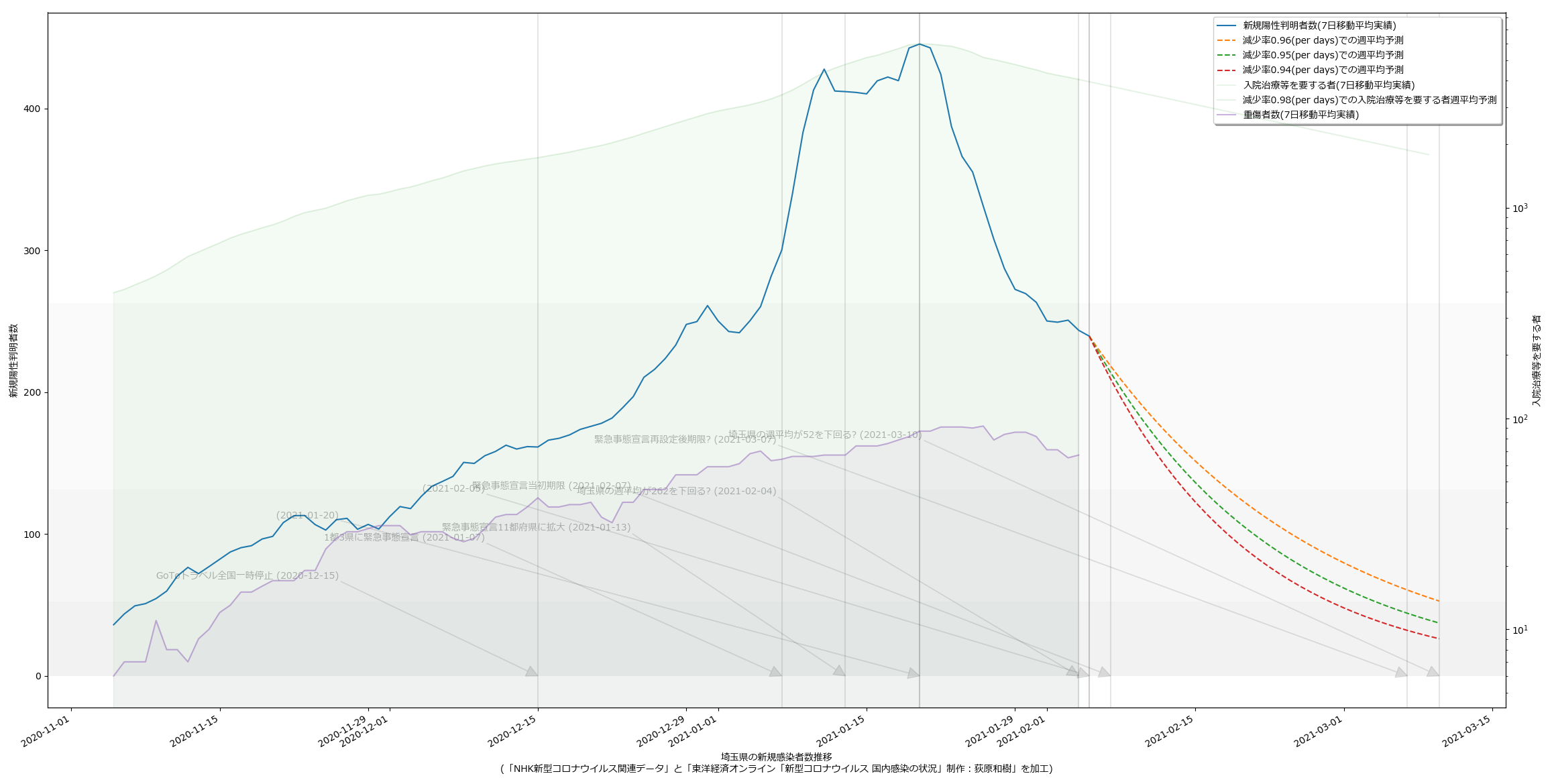
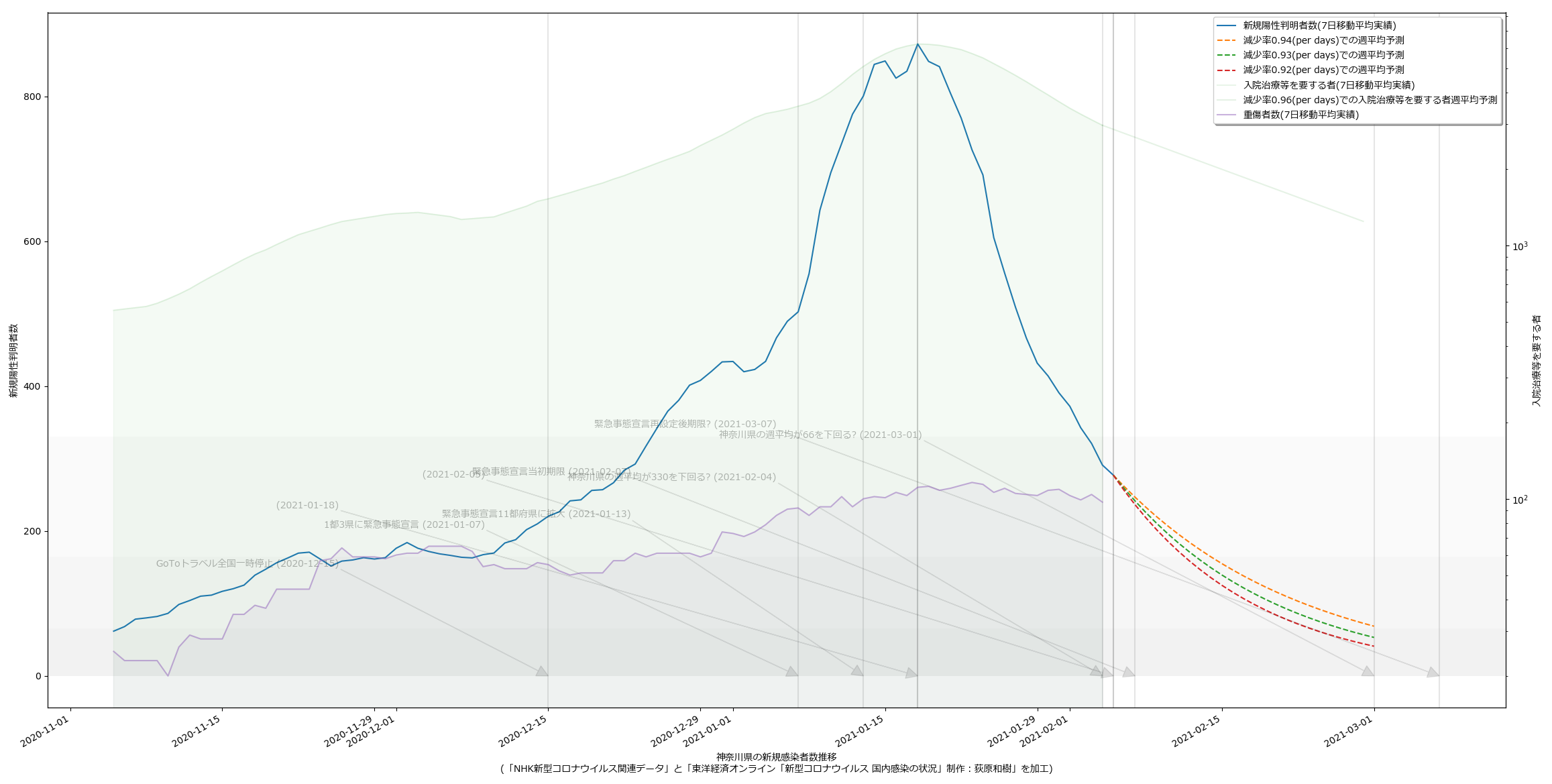
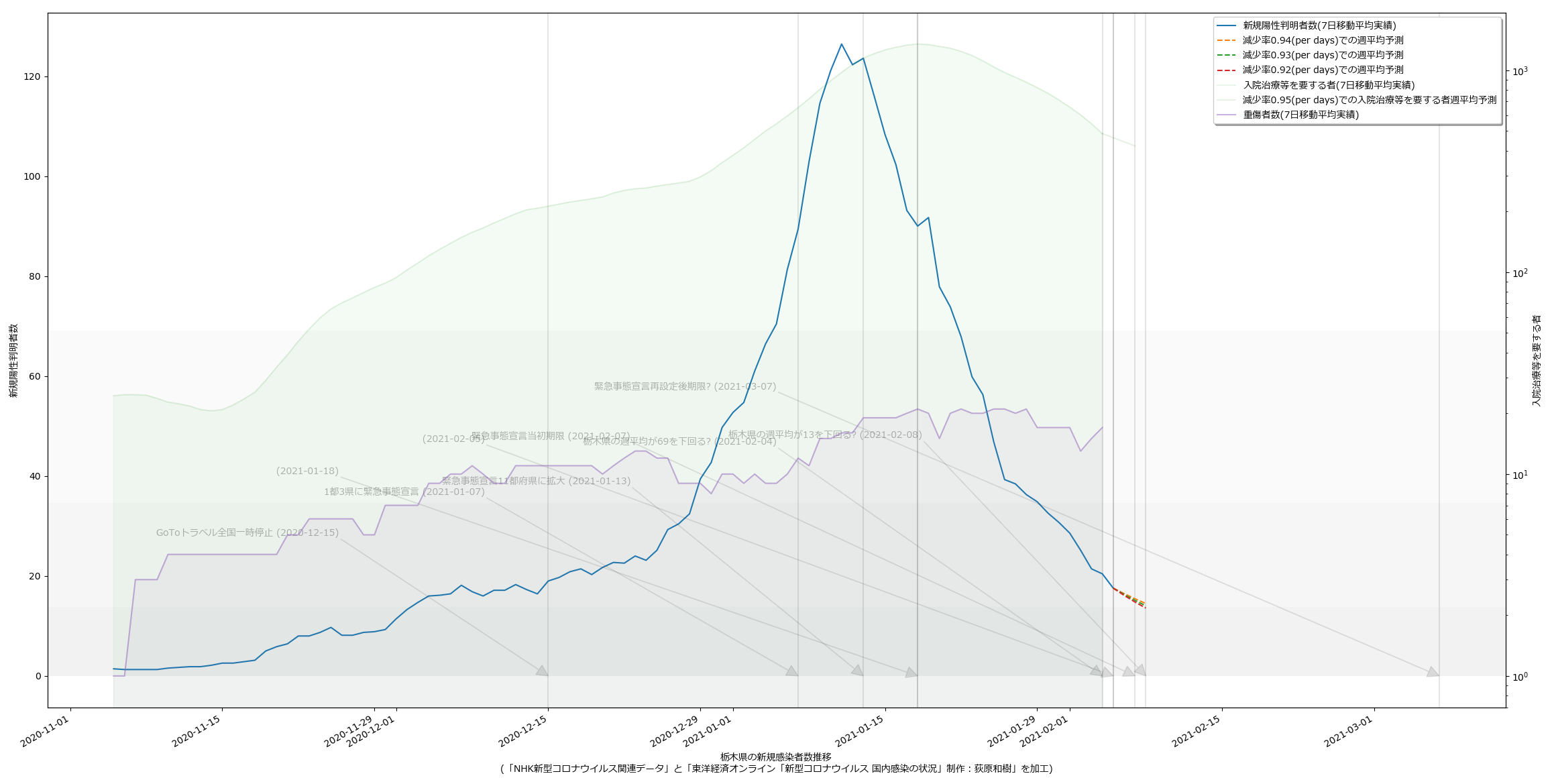
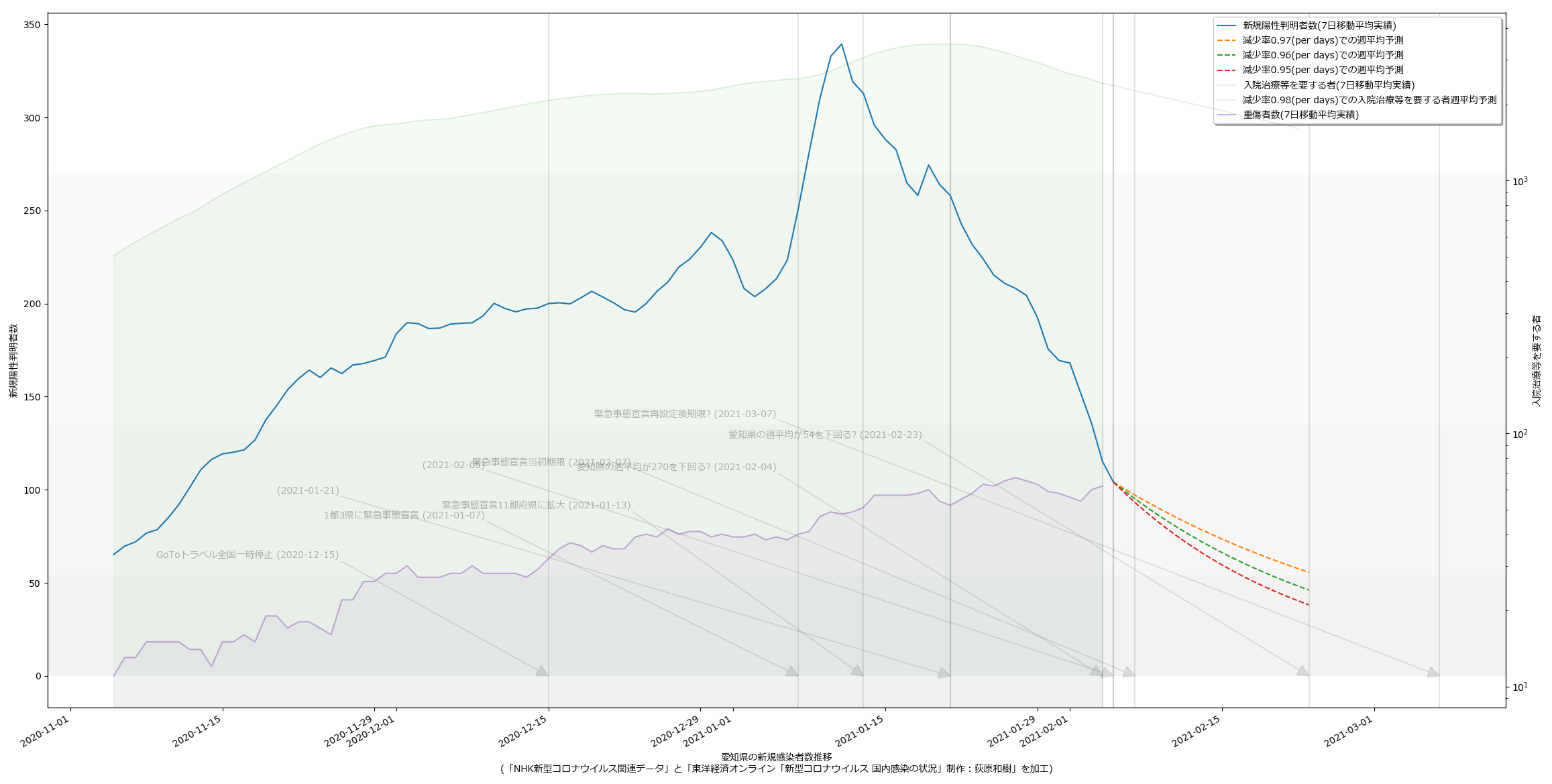
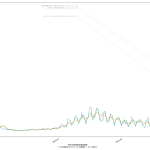
10都道府県で宣言延長が決定し、また、栃木で解除されることを受けて、人口比の考え方を取り込み、今問題になっている都道府県について可視化してみた。こんな:
量が多いのでスクリプトの変更については割愛。
というか、各知事の反応に違和感があるわけよ。特に大阪。ワイドショーのコメンテーターも同じ反応してたが、吉村知事が「300人」と口走ったときには絶句した。これはかなりマズいのではないか。西村大臣の500は「なんも考えてません」宣言に等しいアホ回答だったが、吉村知事の場合はたぶん違う。ちゃんと理解して言ってる。だからこそマズい。ワタシのアホ予測で、63人(東京で100人相当)を下回るには4月5日まで待たなければならず、それは非常によろしくない、ということに基づいて政治判断をしたい、ということならわかるのだが、それ以前に、重症者数がいまだ減少に転じていない。これをもって「300人を下回れば」と言っているのなら、やはり見るべきものを見ていない。大村知事はこれまでの問題発言等々により、個人的には大嫌いだが、少なくとも今回の発言「入院数が~を下回らないと難しい」は、的を射ていると思う。
繰り返すけれども、やはり「見える化の共有」という現代的な道具を使ってコンセンサスを作っていく、というのがメディアの大事な役割であり、そしてテレビはその力で「吉村知事を動かす」ことが出来るんだと思うんだ。「重傷者が減っていない」という客観的事実が「考え方の違い」であってはならない。というか見てる人と見てない人がいる、という状況があってはいけない。と思う。



![[matplotlib] 続・「ヒストグラムの推移」を延々並べるわけにもいかんので](http://hhsprings.pinoko.jp/site-hhs/wp-content/uploads/2018/01/img_5a539d144c52f-150x150.png)
![[matplotlib] 「ヒストグラムの推移」を延々並べるわけにもいかんので](http://hhsprings.pinoko.jp/site-hhs/wp-content/uploads/2017/12/img_5a47b8edd586c-150x150.png)