(11)に引き続き、どちらかといえば「整理」ではあるんだけれど、というよりは、「データ保守」のお助け道具がそろそろ欲しいな、てやつ。このシリーズ全体が「遊び」に徹する都合、なぜか Basemap ネタにも行き着く、の巻。
まずは最初に、これを仕込んでしまいたい。領域をあんまり厳密に決めずに処理した場合でも、「ぴったり陸域が収ま」って欲しい、いつでも。
なんかこれだけのために一個モジュール作るのもなんだなぁ、とも思いつつも、get_bound だけ収めたモジュールを作っておく:
1 # -*- coding: utf-8 -*-
2 import numpy as np
3
4
5 def get_bound(arr, f=np.isnan):
6 #
7 def _gb(arr, axis, f):
8 _sl = [slice(None), ] * arr.ndim
9 idx_l = 0
10 for i in range(arr.shape[axis]):
11 idx_l = i
12 _sl[axis] = slice(i, i + 1)
13 if not np.all(f(arr[_sl])):
14 break
15 idx_h = arr.shape[axis]
16 for i in range(arr.shape[axis], 1, -1):
17 idx_h = i
18 _sl[axis] = slice(i - 1, i)
19 if not np.all(f(arr[_sl])):
20 break
21 return slice(idx_l, idx_h)
22 #
23 return [_gb(arr, axis, f) for axis in range(arr.ndim)]
結果 converted_loader からだけでなく使うことになったので、まぁ分けたのは正解だったが、これはのちほど。で、converted_loader.py に get_bound を仕込む:
1 # -*- coding: utf-8 -*-
2 import os
3 import logging
4 try:
5 # for CPython 2.7
6 import cPickle as pickle
7 except ImportError:
8 # Python 3.x or not CPython
9 import pickle
10 import numpy as np
11 import scipy.ndimage as ndimage
12 import np_utils
13
14
15 _logger = logging.getLogger(__name__)
16
17
18 def _calc_pos0(lon_or_lat, lseg):
19 a = (lon_or_lat % 100) / lseg
20 b = (a - int(a)) / (1 / 8.)
21 c = (b / (1 / 10.)) % 10
22 return (
23 int(a),
24 int(b),
25 int(c),
26 round((int(a) + int(b) / 8. + int(c) / 8. / 10.) * lseg, 9),
27 round((int(a) + int(b) / 8. + (int(c) + 1) / 8. / 10.) * lseg, 9))
28
29
30 def _calc_from_latlon(lat, lon):
31 _fromlat = _calc_pos0(lat, 2/3.)
32 _fromlon = _calc_pos0(lon, 1.)
33 return (
34 "{}{}/{}{}/{}{}".format(
35 _fromlat[0], _fromlon[0],
36 _fromlat[1], _fromlon[1],
37 _fromlat[2], _fromlon[2]),
38 (_fromlat[3], _fromlon[3] + 100),
39 (_fromlat[4], _fromlon[4] + 100))
40
41
42 _shape = (150, 225)
43
44
45 def _load0(fn):
46 with open(fn, "rb") as fi:
47 _d = pickle.load(fi)
48 _st = _d["startPoint"]
49 _raw = _d["elevations"]
50 elevs = np.full((_shape[1] * _shape[0], ), np.nan)
51 elevs[_st:len(_raw) + _st] = _raw
52 elevs = elevs.reshape((_shape[0], _shape[1]))
53 return np.flipud(elevs)
54
55
56 def load(lat, lon, convert_fun=None, use_dem10=True):
57 base, lc, uc = _calc_from_latlon(lat, lon)
58
59 dem05fn = "__converted/DEM05/" + base + ".data"
60 dem10fn = "__converted/DEM10/" + base + ".data"
61
62 elevs1, elevs2 = None, None
63
64 if use_dem10 and os.path.exists(dem10fn):
65 elevs1 = _load0(dem10fn)
66 if convert_fun:
67 elevs1 = convert_fun(elevs1)
68
69 if os.path.exists(dem05fn):
70 elevs2 = _load0(dem05fn)
71 if convert_fun:
72 elevs2 = convert_fun(elevs2)
73
74 if elevs1 is None:
75 if elevs2 is None:
76 elevs = np.full((_shape[0], _shape[1]), np.nan)
77 if convert_fun:
78 elevs = convert_fun(elevs)
79 _logger.info("data was not found: {}".format(base))
80 else:
81 elevs = elevs2
82 _logger.info("DEM5: {}".format(base))
83 else:
84 elevs = elevs1
85 if elevs2 is not None:
86 notnan = (np.isnan(elevs2) == False)
87 elevs[notnan] = elevs2[notnan]
88 _logger.info("DEM5 + DEM10: {}".format(base))
89 else:
90 _logger.info("DEM10: {}".format(base))
91
92 return elevs, lc, uc
93
94
95 def _calc_cellnums(lc_lat, lc_lon, uc_lat, uc_lon):
96 #
97 ydis = (2. / 3 / 8 / 10)
98 xdis = (1. / 8 / 10)
99
100 # adjust startpoint to center of mesh
101 # (for special boundary case.)
102 lc_lat = int(lc_lat / ydis) * ydis + ydis / 2.
103 lc_lon = int(lc_lon / xdis) * xdis + xdis / 2.
104
105 ycells = int(np.ceil(uc_lat / ydis) - np.floor(lc_lat / ydis))
106 xcells = int(np.ceil(uc_lon / xdis) - np.floor(lc_lon / xdis))
107
108 return xdis, ydis, xcells, ycells, lc_lat, lc_lon
109
110 def _load_range0(lc_lat, lc_lon, uc_lat, uc_lon, convert_fun=None, use_dem10=True):
111 #
112 def _stfun(fun, left, right):
113 if left is None:
114 return right
115 return fun((left, right))
116 #
117 xdis, ydis, xcells, ycells, lc_lat, lc_lon = \
118 _calc_cellnums(lc_lat, lc_lon, uc_lat, uc_lon)
119
120 result, lc, uc = None, None, None
121 #
122 for i in range(ycells):
123 tmp = None
124 for j in range(xcells):
125 e, lc_tmp, uc_tmp = load(
126 lc_lat + ydis * i,
127 lc_lon + xdis * j,
128 convert_fun, use_dem10)
129 #
130 if not lc:
131 lc = lc_tmp
132 uc = uc_tmp
133 #
134 tmp = _stfun(np.hstack, tmp, e)
135 #
136 result = _stfun(np.vstack, result, tmp)
137 #
138 return result, lc, uc
139
140
141 def _common_divisors(a, b):
142 while b:
143 a, b = b, a % b
144 return [d for d in range(2, a // 2 + 1) if a % d == 0] + [a]
145
146
147 class _DownSizer(object):
148 def __init__(self, level):
149 self.level = level
150
151 def __call__(self, d):
152 if self.level == 1:
153 return d
154 return ndimage.zoom(
155 d,
156 1. / self.level,
157 mode="nearest",
158 prefilter=False)
159
160
161 def load_range(
162 lat1, lon1, lat2, lon2,
163 use_dem10=True):
164
165 lc_lat = min(lat1, lat2)
166 lc_lon = min(lon1, lon2)
167 uc_lat = max(lat1, lat2)
168 uc_lon = max(lon1, lon2)
169
170 #
171 thr = 2000 * 2000
172 #
173 dummy1, dummy2, xcells, ycells, dummy3, dummy4 = \
174 _calc_cellnums(lc_lat, lc_lon, uc_lat, uc_lon)
175 level = 1
176 cell_sizer = _DownSizer(level)
177 whole_sizer = _DownSizer(level)
178 cdivs0 = [1] + _common_divisors(_shape[0], _shape[1])
179 cdivs1 = [1] + _common_divisors(
180 _shape[0] * ycells, _shape[1] * xcells)
181 for lev in cdivs1:
182 if (_shape[0] * ycells * _shape[1] * xcells) / (lev * lev) > thr:
183 level = lev
184 for lev in cdivs0:
185 if lev <= level:
186 cell_sizer = _DownSizer(lev)
187 whole_sizer = _DownSizer(level / lev)
188 #
189 ms = 5 * cell_sizer.level * whole_sizer.level
190 _logger.info("mesh size: 5m x ({} x {}) = {}m".format(
191 cell_sizer.level,
192 whole_sizer.level,
193 ms))
194 #
195 elevs, lc, uc = _load_range0(
196 lc_lat, lc_lon, uc_lat, uc_lon,
197 convert_fun=cell_sizer, use_dem10=use_dem10)
198 #
199 elevs = whole_sizer(elevs)
200 _logger.info("shape before clip: (%d, %d)" % elevs.shape)
201
202 X = np.linspace(lc[1], uc[1], elevs.shape[1])
203 Y = np.linspace(lc[0], uc[0], elevs.shape[0])
204 xmin, xmax = np.where(X <= lc_lon)[0][-1], np.where(X >= uc_lon)[0][0]
205 ymin, ymax = np.where(Y <= lc_lat)[0][-1], np.where(Y >= uc_lat)[0][0]
206 elevs = elevs[ymin:ymax, xmin:xmax]
207 X, Y = X[xmin:xmax], Y[ymin:ymax]
208 #
209 bd = np_utils.get_bound(elevs)
210 elevs = elevs[bd]
211 X, Y = X[bd[1]], Y[bd[0]]
212 #
213 _logger.info("shape after clip: (%d, %d)" % elevs.shape)
214 _logger.info("area size (aprx.): W-E: %dm, S-N: %dm" % (
215 elevs.shape[1] * ms,
216 elevs.shape[0] * ms))
217
218 return X, Y, elevs
converted_loader については今回はこれだけ。
で、「保守のお助け」なんだけれどもね。だんだん持ってるデータが増えてくるに従って、「あら、あたし、どれを持ってて、どれを持ってないのかしら?」てのがわかりにくくなってきたのですよ。特にね、「DEM05は持ってるが、DEM10 で補わなければならないのはどこか」を素早く知るのが難しくなってきてるの。さすがにファイル名規則だけから絵を想像するのはむつかしいし、標高可視化を「やってみて、ログから抜けを探す」の、手間過ぎるし。だからこれはもう、「持ってるデータの可視化(見える化)」が欲しかろうぞ、ってな。
まずおさらい。「ダウンロード→展開→「オレオレ構造に converter.py で変換」」を、ワタシがどう配置してるか。こんなね:
1 me@host: FG-GML$ find __arch
2 __arch
3 __arch/FG-GML-4839-56-DEM5A.tar.bz2
4 ...
5 __arch/FG-GML-5238-66-DEM5B.tar.bz2
6 __arch/FG-GML-5238-67-DEM10A.tar.bz2
7 __arch/FG-GML-5238-67-DEM10B.tar.bz2
8 __arch/FG-GML-5238-67-DEM5A.tar.bz2
9 ...
zipじゃなくtar.bz2になってるのはこれ。
1 me@host: FG-GML$ find __extracted
2 __extracted
3 __extracted/FG-GML-5339-00-DEM5A
4 __extracted/FG-GML-5339-00-DEM5A/FG-GML-5339-00-00-DEM5A-20130702.xml
5 __extracted/FG-GML-5339-00-DEM5A/FG-GML-5339-00-01-DEM5A-20130702.xml
6 __extracted/FG-GML-5339-00-DEM5A/FG-GML-5339-00-02-DEM5A-20130702.xml
7 ...
8 __extracted/FG-GML-5339-00-DEM5A/FG-GML-5339-00-99-DEM5A-20130702.xml
9 ...
10 __extracted/FG-GML-5339-50-dem10b-20090201.xml
11 ...
1 me@host: FG-GML$ find __converted
2 __converted
3 __converted/DEM05
4 __converted/DEM05/4839
5 __converted/DEM05/4839/56
6 __converted/DEM05/4839/56/30.data
7 ...
8 __converted/DEM05/4839/56/61.data
9 ...
10 __converted/DEM10
11 __converted/DEM10/5238
12 __converted/DEM10/5238/57
13 __converted/DEM10/5238/57/00.data
14 __converted/DEM10/5238/57/01.data
15 ...
どれも「オレ様流」なんであって、why言わないように。
さて。「どれさまをダウンロード済みか」の可視化のための第一歩は、converted_loader や converter がやってるファイルの渡り歩きと、ファイル名からの情報お取り寄せを切り出さねばならぬ。ので、こんなのを書いた:
1 # -*- coding: utf-8 -*-
2 import os
3 import re
4
5
6 _FNRGX1 = re.compile(
7 r"FG-GML-(.*)-(.*)-(.*)-DEM5[AB]-.*", flags=re.I)
8 _FNRGX2 = re.compile(
9 r"FG-GML-(.*)-(.*)-DEM10[AB]-.*", flags=re.I)
10 _FNRGX3 = re.compile(
11 r"FG-GML-(.*)-(.*)-DEM([015]*)[AB].*", flags=re.I)
12
13
14 def info_from_filename(fpth_noext, ty):
15 bn = os.path.basename(fpth_noext)
16 if ty == "__extracted":
17 m = _FNRGX1.match(bn)
18 if m: # DEM5 xml
19 return "DEM05", m.group(1), m.group(2), m.group(3)
20 m = _FNRGX2.match(bn)
21 if m: # DEM10 xml
22 return "DEM10", m.group(1), m.group(2), None
23 elif ty == "__arch":
24 m = _FNRGX3.match(bn)
25 return "DEM%02d" % int(m.group(3)), m.group(1), m.group(2), None
26 else:
27 mn2 = os.path.dirname(fpth_noext)
28 mn1 = os.path.dirname(mn2)
29 dty = os.path.basename(os.path.dirname(mn1))
30 return dty, os.path.basename(mn1), os.path.basename(mn2), bn
31
32
33 def walk(ty, meshnum_parse=True):
34 metas = {
35 "__converted": {
36 "exts": (".data",),
37 },
38 "__extracted": {
39 "exts": (".xml",),
40 },
41 "__arch": {
42 "exts": (".zip", ".bz2",),
43 },
44 }
45 tt = lambda s, idx: map(int, (s[:idx], s[idx:])) if s else None
46 meta = metas[ty]
47 for root, dirs, files in os.walk(ty):
48 for fn in files:
49 base, ext = os.path.splitext(fn)
50 if ext not in meta["exts"]:
51 continue
52 fpth_noext = os.path.join(root, base)
53 dty, mn1, mn2, mn3 = info_from_filename(fpth_noext, ty)
54 if meshnum_parse:
55 mesh1idx = tt(mn1, 2)
56 mesh2idx = tt(mn2, 1)
57 mesh3idx = tt(mn3, 1)
58 yield fpth_noext + ext, dty, mesh1idx, mesh2idx, mesh3idx
59 else:
60 yield fpth_noext + ext, dty, mn1, mn2, mn3
これは converter で使えて、「「どれさまをダウンロード済みか」の可視化」にも使える「渡り歩き走りたい」を狙ったもの。
まずこれで converter.py を書き換えておく:
1 # -*- coding: utf-8 -*-
2 import re
3 import os
4 try:
5 # for CPython 2.7
6 import cPickle as pickle
7 except ImportError:
8 # Python 3.x or not CPython
9 import pickle
10 from os import path
11 from distutils.dir_util import create_tree
12 import numpy as np
13 import datafile_tree
14
15
16 def _dump_data(
17 st, _raw,
18 mfirst, msecond, mthird=None):
19
20 def _pickle_dump(data, dest_dir, dest_filebase):
21 fn = path.join(
22 dest_dir, "{}.data".format(dest_filebase))
23 if all(np.isnan(data["elevations"])):
24 return
25 pickle.dump(
26 data,
27 open(fn, "wb"),
28 protocol=-1)
29 print(fn, data["startPoint"], len(data["elevations"]))
30
31 if mthird: # DEM5
32 dest_dir_top = "__converted/DEM05"
33 else:
34 dest_dir_top = "__converted/DEM10"
35 dest_dir = path.join(dest_dir_top, mfirst, msecond)
36 create_tree(dest_dir, ["dummy"])
37 if mthird: # DEM5
38 data = {"startPoint": st, "elevations": _raw}
39 _pickle_dump(data, dest_dir, mthird)
40 else: # DEM10
41 shape = (750, 1125)
42 tmp = np.full((shape[1] * shape[0], ), np.nan)
43 tmp[st:len(_raw) + st] = _raw
44 tmp = np.flipud(tmp.reshape((shape[0], shape[1])))
45 dy = shape[0] / 5
46 dx = shape[1] / 5
47 for y in range(5):
48 for x in range(5):
49 data22 = tmp[
50 y * dy:(y + 1) * dy,
51 x * dx:(x + 1) * dx]
52 # zoom 2x2 with kronecker product
53 data = np.kron(data22, np.ones((2, 2)))
54 # split into DEM5 level
55 dy0 = data.shape[0] / 2
56 dx0 = data.shape[1] / 2
57 for y0 in range(2):
58 for x0 in range(2):
59 leaf = data[
60 y0 * dy0:(y0 + 1) * dy0,
61 x0 * dx0:(x0 + 1) * dx0]
62 leaf = np.flipud(leaf).reshape(
63 leaf.shape[0] * leaf.shape[1])
64 idx = np.where(np.isnan(leaf) == False)[0]
65 if len(idx) == 0:
66 continue
67 dest_filebase = "{}{}".format(
68 y * 2 + y0,
69 x * 2 + x0)
70 _pickle_dump(
71 {
72 "startPoint":
73 int(idx[0]),
74 "elevations":
75 leaf[idx[0]:idx[-1] + 1].tolist()
76 },
77 dest_dir,
78 dest_filebase)
79
80
81 _STRGX = re.compile(
82 r"<gml:startPoint> *(.*) (.*)</gml:startPoint>")
83
84
85 def _convert_xml(srcgml, dty, mfirst, msecond, mthird):
86 _st = (0, 0)
87 _elevs_raw = []
88
89 with open(srcgml, "r") as fi:
90 # find start of grid data
91 while True:
92 line = fi.readline()
93 if "gml:tupleList" in line:
94 break
95
96 # read grid data
97 while True:
98 line = fi.readline().strip()
99 if not line:
100 continue
101 if "gml:tupleList" in line:
102 break
103 ty, d = line.split(",")
104 d = float(d)
105 # we need to treat sea level always NaN rather than zero.
106 if ty.decode('cp932') in (u'海水面', u'データなし'):
107 d = float('NaN')
108 # invalid data are stored with '-9999.0'
109 elif d == -9999.0:
110 d = float('NaN')
111 _elevs_raw.append(d)
112
113 # read start-point
114 while True:
115 line = fi.readline()
116 m = _STRGX.search(line)
117 if m:
118 _st = (
119 int(m.group(1)), int(m.group(2)))
120 break
121
122 # pickle data
123 if dty == "DEM05": # DEM5
124 shape = (150, 225)
125 st = _st[1] * shape[1] + _st[0]
126 _dump_data(
127 st, _elevs_raw, mfirst, msecond, mthird)
128 else: # DEM10
129 shape = (750, 1125)
130 st = _st[1] * shape[1] + _st[0]
131 _dump_data(st, _elevs_raw, mfirst, msecond)
132
133 #
134 for srcgml, dty, mesh1idx, mesh2idx, mesh3idx in \
135 datafile_tree.walk("__extracted", meshnum_parse=False):
136 _convert_xml(srcgml, dty, mesh1idx, mesh2idx, mesh3idx)
これまでやってきたもので確認して、問題ないので、これでよしとする。
さて。「「どれさまをダウンロード済みか」の可視化」。簡単にいえば「(元の)GML 一個」が要素一個に対応する行列を可視化するだけ。ただし、「まだまだ持っていないものが多い」場合、どこが抜けているのか、単に matplotlib の imshow や pcolor するだけでは良くわからないのね。ので、Basemap と組み合わせて、海岸線と一緒に表示してやろう、と。
出来上がったものは別に難しくない:
1 # -*- coding: utf-8 -*-
2 import os
3 import re
4 import numpy as np
5 import np_utils
6
7
8 # ##################################################
9 #
10 import datafile_tree
11
12
13 _all = np.zeros((135 * 8 * 10, 80 * 8 * 10))
14 for fn, dty, m1, m2, m3 in datafile_tree.walk("__converted"):
15 y = m1[0] * 8 * 10 + m2[0] * 10 + m3[0]
16 x = m1[1] * 8 * 10 + m2[1] * 10 + m3[1]
17 print(dty, m1, m2, m3, (y, x))
18 _all[y, x] += 2. if dty == "DEM05" else 1.
19
20 bd = np_utils.get_bound(_all, f=lambda a: a == 0.)
21 _x2lon = lambda x: x / (8 * 10.) + 100.
22 _y2lat = lambda y: y / (8 * 10.) * (2 / 3.)
23 elevs = _all[bd]
24 X = np.linspace(
25 _x2lon(bd[1].start),
26 _x2lon(bd[1].stop - 1),
27 elevs.shape[1])
28 Y = np.linspace(
29 _y2lat(bd[0].start),
30 _y2lat(bd[0].stop - 1),
31 elevs.shape[0])
32
33
34 # ##################################################
35 #
36 from mpl_toolkits.basemap import Basemap
37 import matplotlib.pyplot as plt
38 import matplotlib.cm as cm
39
40 fig = plt.figure()
41 fig.set_size_inches(16.53 * 2, 11.69 * 2)
42 m = Basemap(
43 llcrnrlon=X[0],
44 llcrnrlat=Y[0],
45 urcrnrlon=X[-1],
46 urcrnrlat=Y[-1],
47 resolution='f',
48 projection='merc')
49
50 lons, lats = np.meshgrid(X, Y)
51 x, y = m(lons, lats)
52
53 m.drawcoastlines(color='0.9', linewidth=1)
54 #m.fillcontinents(alpha=0.3)
55 circles = np.arange(10, 90, 0.5)
56 m.drawparallels(circles, labels=[1, 1, 1, 1])
57 meridians = np.arange(-180, 180, 0.5)
58 m.drawmeridians(meridians, labels=[1, 1, 1, 1])
59 m.pcolor(x, y, elevs, cmap=cm.summer)
60 plt.savefig("motterumotteru.png", bbox_inches="tight")
61 #plt.show()
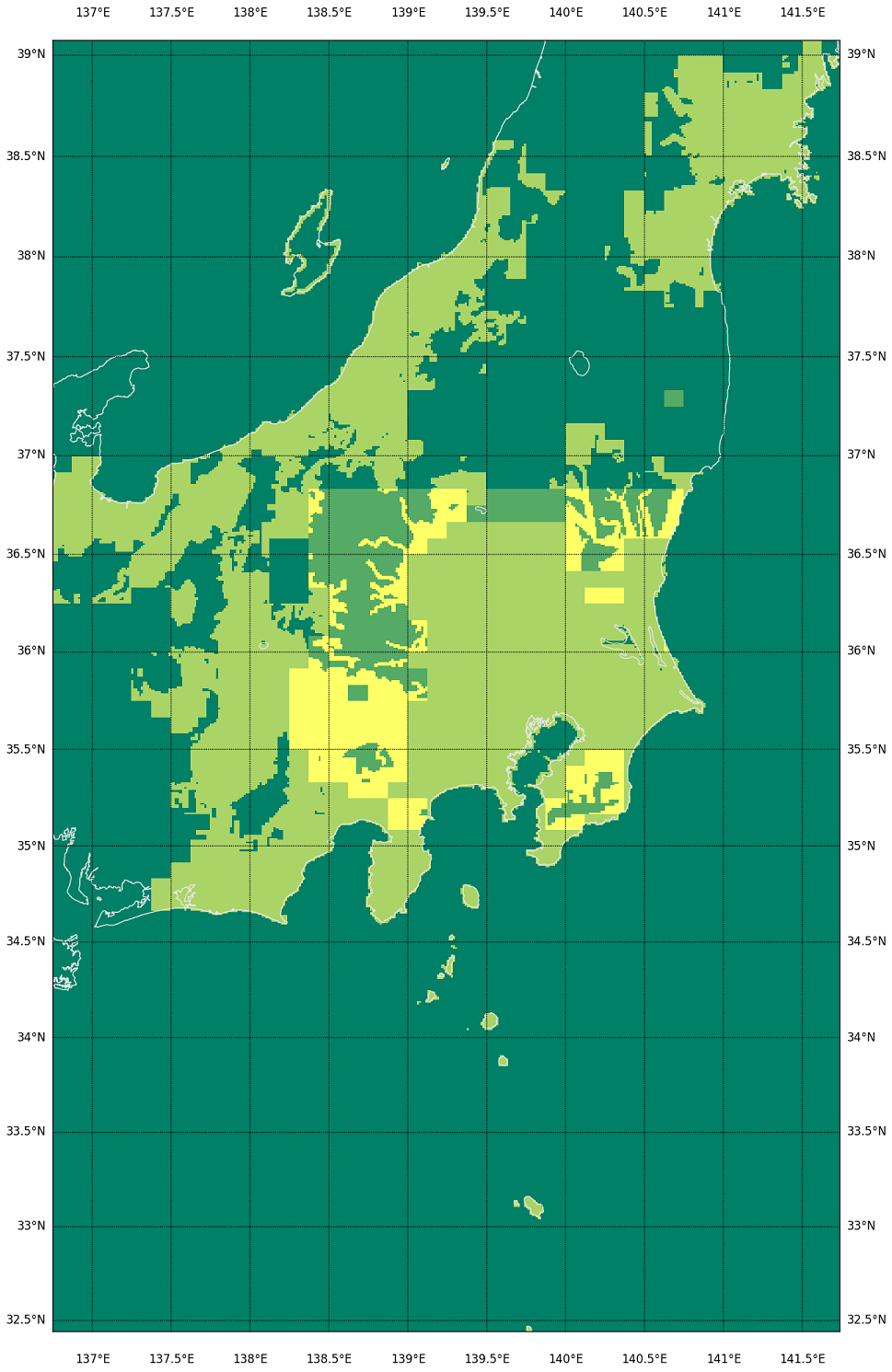
ハイライトした「_all」という行列が、「もとのXML一個(厳密にはDEM05レベルのセル)」に直接対応するもので、上で作った datafile_tree を使って渡り歩いて、DEM05 がある場所を +2、DEM10 がある場所を +1 している。つまり両方持っている場所は 3 になる。これを最後に Basemap とともに表示している。
あ、上で「のちほど」と言った np_utils.get_bound も使ってます。
出来上がったものが単純だからといって簡単だったということにはならないわけで。久しぶりに使ったこともあるし、Basemap ってなんだかんだ、とっつき悪いところもあるので、ちょいちょい苦労はしながら作った。
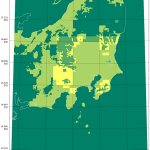
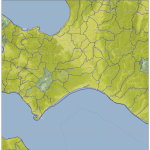
てわけで、今持ってるのをコヤツで可視化するとこんななの:
うん、わかりすい。この処理は遅いけど、必要なときにしか動かさないんで、まぁいいだろう。メッシュ番号との対応がわかるとなお良いのだが、まぁなくても絵(この絵)と絵(基盤地図情報ダウンロードページの地図からの選択)の比較でわからんこともないから。
濃い緑が「データがない」とこ、黄緑が「DEM05だけ」、薄い緑が「DEM10だけ」、黄色が「DEM05とDEM10両方」のとこ。DEM10 がある場所は要するに「DEM05の欠落」が問題になって、DEM10 を後から取得した場所ね。で、陸域で大きな矩形ではない「滑らかな抜け」部分が、「DEM05をダウンロードして持っているのに欠落している」部分。(全体を持ってるはずの新潟と、持ってないはずの愛知を比較してみて。)
海岸線がない場合、例えば今の例だと茨城・福島の部分の抜けがわかりにくくなるのよ。飛び地で取得すればするほどそういう具合になる。
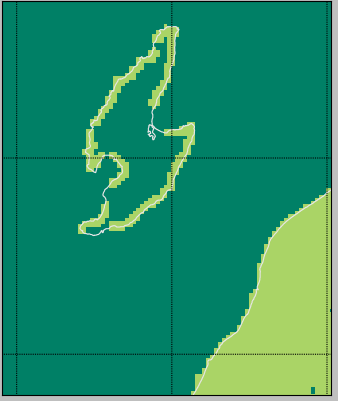
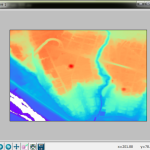
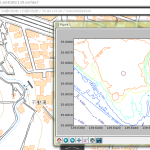
「DEM05の欠落」なんだけどさ、こうやってやってみて改めてわかったんだけど、ほんと「大事なとこ以外はぞんざい」なのね。上で貼り付けた絵だと解像度の関係でわかりにくいんだけれど、佐渡島もヒドイの。ここだけ拡大したのがこれ:
海岸線だけ、なのな。
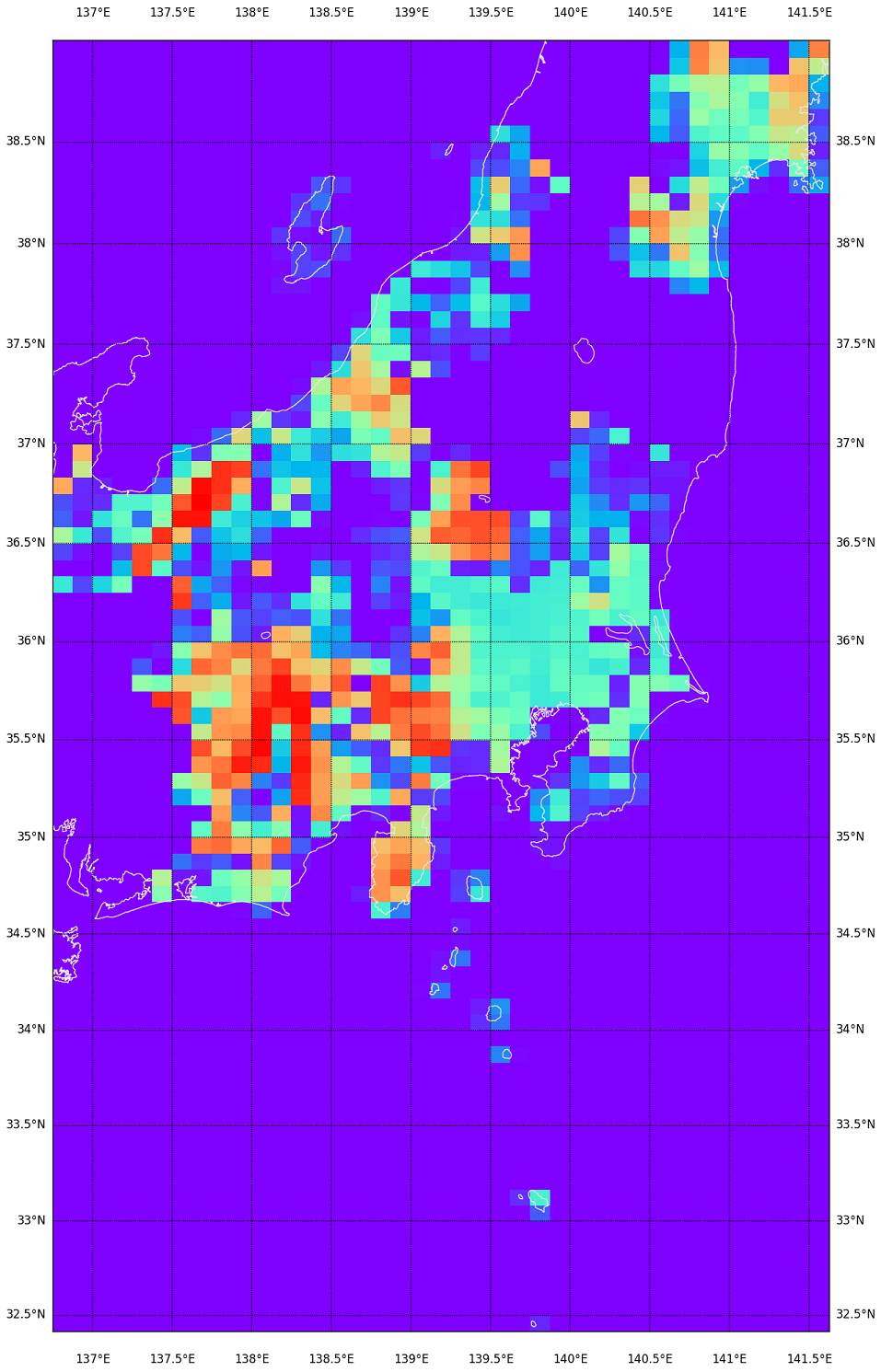
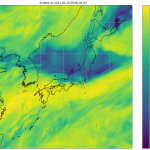
さて、「詳細に知る」にはこれでいいんだけど、いかんせん若干処理が重いので、先に DEM05 の抜け具合だけ軽く知りたかったりもする。ダウンロードしたアーカイブのファイルサイズで可視化、も良さそうだ:
1 # -*- coding: utf-8 -*-
2 import os
3 import re
4 import numpy as np
5 import np_utils
6
7
8 # ##################################################
9 #
10 import datafile_tree
11
12
13 _all = np.zeros((135 * 8, 80 * 8))
14 for fn, dty, m1, m2, m3 in datafile_tree.walk("__arch"):
15 if dty == "DEM05":
16 y = m1[0] * 8 + m2[0]
17 x = m1[1] * 8 + m2[1]
18 print(dty, m1, m2, (y, x))
19 _all[y, x] = os.stat(fn).st_size
20
21 bd = np_utils.get_bound(_all, f=lambda a: a == 0.)
22 _x2lon = lambda x: x / 8. + 100.
23 _y2lat = lambda y: y / 8. * (2 / 3.)
24 elevs = _all[bd]
25 X = np.linspace(
26 _x2lon(bd[1].start),
27 _x2lon(bd[1].stop - 1),
28 elevs.shape[1])
29 Y = np.linspace(
30 _y2lat(bd[0].start),
31 _y2lat(bd[0].stop - 1),
32 elevs.shape[0])
33
34
35 # ##################################################
36 #
37 from mpl_toolkits.basemap import Basemap
38 import matplotlib.pyplot as plt
39 import matplotlib.cm as cm
40
41 fig = plt.figure()
42 fig.set_size_inches(16.53 * 2, 11.69 * 2)
43 m = Basemap(
44 llcrnrlon=X[0],
45 llcrnrlat=Y[0],
46 urcrnrlon=X[-1],
47 urcrnrlat=Y[-1],
48 resolution='f',
49 projection='merc')
50
51 lons, lats = np.meshgrid(X, Y)
52 x, y = m(lons, lats)
53
54 m.drawcoastlines(color='0.9', linewidth=1)
55 #m.fillcontinents(alpha=0.3)
56 circles = np.arange(10, 90, 0.5)
57 m.drawparallels(circles, labels=[1, 1, 1, 1])
58 meridians = np.arange(-180, 180, 0.5)
59 m.drawmeridians(meridians, labels=[1, 1, 1, 1])
60 m.pcolor(x, y, elevs, cmap=cm.rainbow)
61 plt.savefig("motterumotteru-rough.png", bbox_inches="tight")
62 #plt.show()
かなり処理が軽い。のでこれがわかりやすければ、先にこれで見るのは手だが…?
カラーマップは summer だといまひとつだったんで、rainbow。寒色ほどサイズが小さいということ。海岸を含む領域が寒色になるのは当たり前。 でもこれは DEM05 の欠落具合は表現出来ないなぁ…。XML のサイズがどうも標高が高い領域が大きくなるみたいで、そちらの傾向の方が強く出てしまってるようだ。ファイルサイズの大きい赤い部分ほど、抜けだらけだったとこだもん。この視覚化はあんまり役に立たないか…。(でも持ってる持ってないはわかりやすいか。)
ちなみに、もしもはじめて Basemap を知ったという人で、興味を持って始めてみようと思う人には最初の注意。上で使ってる「resolution=’f’」ですが、これは海岸線(など)の細かさを制御するもの、なのですが、最初から「’f’」でやらないこと。とにかく色々試行錯誤してる間は「resolution=’c’」でやってください。処理時間が全然違うのよ。’f’は「full」って意味。これ、30秒とか平気でかかる。「’c’」は「crude」、つまり「ぞんざい」。これは(‘h’や’f’に較べて)猛烈に速く、また、日本全国、のような広域の場合はこれでも実用になる。
2021-05-07追記:
紹介した basemap だが、作者である Jeff Whitaker が「basemap プロジェクト」としての保守を完全にやめ、pip でのインストールも不可能になった。自身による説明:
Deprecation Notice
Basemap is deprecated in favor of the Cartopy project. See notes in Cartopy, New Management, and EoL Announcement for more details.
リンク先をちょっと読むに、「より大きなプロジェクトに取り込まれた」ということに見える。cartopy はワタシ自身にとって完全に未知なのでまだ何も言わないけれど、余力があったらそのうち何か書くかも。