(7.1)で気付いた通りなんだけど、だんだん取得データ増やしてきたら、欠落データが思ったよりずっと多いのね。
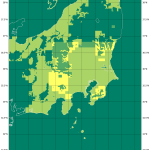
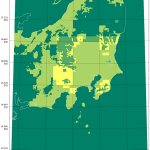
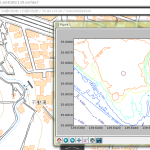
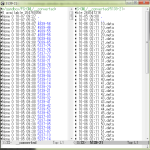
(9)で載せようか迷ってたのは、(9)よりも遥かに広域の「関東平野ほぼ全域」。実際はこの時点で試してたのね。zoom(1/25)で。でもこんななの:
ダウンロードしていないから、という部分は若干あるけれど、「滑らかな欠落部分」があることから、本当に「データ=「データなし」」がこのように分布しているのだということがわかると思う。富士山はやったはずなのに気付かなかったな。なんで山梨が集中的に欠落してるんだろうか。
で、(7.1)で「DEM10ならある、とかないかなぁ?」を観察してみた。
房総半島の「5239-67」を覗いてみたら、あぁ、欠落はなさそうだ。DEM5 の欠落を補うのに DEM10 を使うことは出来そう。更新日付は DEM5 が 2013 年なのに対し DEM10 は 2009 年みたいで、このズレが出る場合もあるかもしれないけど、欠落よりはいいだろう。
まずデータ変換と格納場所をどうしようか。DEM5 だけ扱ってたぶんには「一次メッシュ番号/二次メッシュ番号/三次メッシュ番号.data」で良かった。DEM10 の場合三次メッシュでのファイル分割はなくて、二次メッシュに対応したファイル一個に 1125×750 が含まれている(DEM5のセル一個の5×5倍)。
変換時に頑張るか、ロード時に頑張るか。前者にしたい。つまり DEM10 を三次メッシュレベルに分解して格納してしまいたい。ファイルサイズは増えるけれど、DEM10 は「欠落が問題になる場所でのみ」ダウンロードして使うことしか考えてないので、大問題ではないから。
ファイル名規則とフォルダをどうすっかね? 拡張子を変えて同じ場所に置くか、フォルダで分けてしまうかどっちかなんだけど。フォルダ分けた方が扱いやすいかね? そうしようか。「DEM05/」と「DEM10/」というふうにトップレベルで分けてしまおう。これまで変換済みの DEM5 データの移動もこれなら楽だしね。
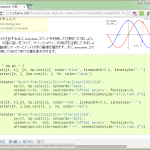
てわけで、変換処理(converter.py)を一気に改造:
1 # -*- coding: utf-8 -*-
2 import re
3 import os
4 try:
5 # for CPython 2.7
6 import cPickle as pickle
7 except ImportError:
8 # Python 3.x or not CPython
9 import pickle
10 from os import path
11 from distutils.dir_util import create_tree
12 import numpy as np
13
14
15 def _dump_data(
16 st, _raw,
17 mfirst, msecond, mthird=None):
18
19 def _pickle_dump(data, dest_dir, dest_filebase):
20 if all(np.isnan(data["elevations"])):
21 return
22 pickle.dump(
23 data,
24 open(
25 path.join(
26 dest_dir, "{}.data".format(dest_filebase)), "wb"),
27 protocol=-1)
28
29 if mthird: # DEM5
30 dest_dir_top = "__converted/DEM05"
31 else:
32 dest_dir_top = "__converted/DEM10"
33 dest_dir = path.join(dest_dir_top, mfirst, msecond)
34 create_tree(dest_dir, ["dummy"])
35 if mthird: # DEM5
36 data = {"startPoint": st, "elevations": _raw}
37 _pickle_dump(data, dest_dir, mthird)
38 else: # DEM10
39 shape = (750, 1125)
40 tmp = np.full((shape[1] * shape[0], ), np.nan)
41 tmp[st:len(_raw) + st] = _raw
42 tmp = np.flipud(tmp.reshape((shape[0], shape[1])))
43 dy = shape[0] / 5
44 dx = shape[1] / 5
45 for y in range(5):
46 for x in range(5):
47 data22 = tmp[
48 y * dy:(y + 1) * dy,
49 x * dx:(x + 1) * dx]
50 # zoom 2x2 with kronecker product
51 data = np.kron(data22, np.ones((2, 2)))
52 # split into DEM5 level
53 dy0 = data.shape[0] / 2
54 dx0 = data.shape[1] / 2
55 for y0 in range(2):
56 for x0 in range(2):
57 leaf = data[
58 y0 * dy0:(y0 + 1) * dy0,
59 x0 * dx0:(x0 + 1) * dx0]
60 leaf = np.flipud(leaf).reshape(
61 leaf.shape[0] * leaf.shape[1])
62 idx = np.where(np.isnan(leaf) == False)[0]
63 if len(idx) == 0:
64 continue
65 dest_filebase = "{}{}".format(
66 y * 2 + y0,
67 x * 2 + x0)
68 _pickle_dump(
69 {
70 "startPoint":
71 int(idx[0]),
72 "elevations":
73 leaf[idx[0]:idx[-1] + 1].tolist()
74 },
75 dest_dir,
76 dest_filebase)
77
78
79 _STRGX = re.compile(
80 r"<gml:startPoint> *(.*) (.*)</gml:startPoint>")
81 _FNRGX1 = re.compile(
82 r"FG-GML-(.*)-(.*)-(.*)-DEM5[AB]-.*.xml", flags=re.I)
83 _FNRGX2 = re.compile(
84 r"FG-GML-(.*)-(.*)-DEM10[AB]-.*.xml", flags=re.I)
85
86
87 def _convert_xml(srcgml):
88 _st = (0, 0)
89 _elevs_raw = []
90
91 with open(srcgml, "r") as fi:
92 # find start of grid data
93 while True:
94 line = fi.readline()
95 if "gml:tupleList" in line:
96 break
97
98 # read grid data
99 while True:
100 line = fi.readline().strip()
101 if not line:
102 continue
103 if "gml:tupleList" in line:
104 break
105 ty, d = line.split(",")
106 d = float(d)
107 # we need to treat sea level always NaN rather than zero.
108 if ty.decode('cp932') in (u'海水面', u'データなし'):
109 d = float('NaN')
110 # invalid data are stored with '-9999.0'
111 elif d == -9999.0:
112 d = float('NaN')
113 _elevs_raw.append(d)
114
115 # read start-point
116 while True:
117 line = fi.readline()
118 m = _STRGX.search(line)
119 if m:
120 _st = (
121 int(m.group(1)), int(m.group(2)))
122 break
123
124 # pickle data
125 m = _FNRGX1.match(path.basename(srcgml))
126 if m: # DEM5
127 shape = (150, 225)
128 st = _st[1] * shape[1] + _st[0]
129 _dump_data(
130 st, _elevs_raw, m.group(1), m.group(2), m.group(3))
131 else: # DEM10
132 shape = (750, 1125)
133 st = _st[1] * shape[1] + _st[0]
134 m = _FNRGX2.match(path.basename(srcgml))
135 _dump_data(st, _elevs_raw, m.group(1), m.group(2))
136
137 #
138 for root, dirs, files in os.walk("__extracted"):
139 for fn in files:
140 if ".xml" in fn:
141 srcgml = path.join(root, fn)
142 _convert_xml(srcgml)
143 print(srcgml)
本質とは関係ない部分での改造としては、lowerCorner、upperCorner、low、high の読み込みをもはややめてる。なんかヘンな空白入ってて処理に失敗したのが理由だけど、絶対使わないからさ、この情報。それと、全域が「海水面,-9999.」の場合はファイル自体存在しないものと思っていたら、DEM5B の中にそんなのがいた。無駄なのでこれは捨てることに。
ハイライトした部分が本題で…、頭使った。知恵熱が出そうだ。最初1次元のままやろうとしたけど、無理があったので、ndarray 任せの方法で。ちょっと面白がって欲しいのはクロネッカー積(np.kron)してる部分ね。ここを ndimage.zoom しても悪くはないんだけれど、生データの解像度以上に滑らかなのはオカシイことなので、np.kron での「拡大」をした。
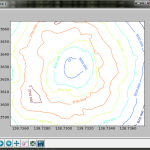
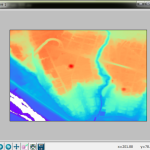
ゴールは「DEM5 にないものを DEM10 から補う」ではあるけれど、先に DEM10 だけで単独で正しくないといけないので確認せねば。単独範囲では形状がわからず、正解かどうかわからなかったので、「5239-66」、「5239-76」、「5239-77」とともに:
オッケーだね。まごうことなき富津岬る。
では、「DEM5 にないものを DEM10 から補う」を。converted_loader.py はこうした:
1 # -*- coding: utf-8 -*-
2 import os
3 try:
4 # for CPython 2.7
5 import cPickle as pickle
6 except ImportError:
7 # Python 3.x or not CPython
8 import pickle
9 import numpy as np
10
11
12 def _calc_pos0(lon_or_lat, lseg):
13 a = (lon_or_lat % 100) / lseg
14 b = (a - int(a)) / (1 / 8.)
15 c = (b / (1 / 10.)) % 10
16 return (
17 int(a),
18 int(b),
19 int(c),
20 round((int(a) + int(b) / 8. + int(c) / 8. / 10.) * lseg, 9),
21 round((int(a) + int(b) / 8. + (int(c) + 1) / 8. / 10.) * lseg, 9))
22
23
24 def _calc_from_latlon(lat, lon):
25 _fromlat = _calc_pos0(lat, 2/3.)
26 _fromlon = _calc_pos0(lon, 1.)
27 return (
28 "{}{}/{}{}/{}{}".format(
29 _fromlat[0], _fromlon[0],
30 _fromlat[1], _fromlon[1],
31 _fromlat[2], _fromlon[2]),
32 (_fromlat[3], _fromlon[3] + 100),
33 (_fromlat[4], _fromlon[4] + 100))
34
35
36 _shape = (150, 225)
37
38
39 def _load0(fn):
40 with open(fn, "rb") as fi:
41 _d = pickle.load(fi)
42 _st = _d["startPoint"]
43 _raw = _d["elevations"]
44 elevs = np.full((_shape[1] * _shape[0], ), np.nan)
45 elevs[_st:len(_raw) + _st] = _raw
46 elevs = elevs.reshape((_shape[0], _shape[1]))
47 return np.flipud(elevs)
48
49
50 def load(lat, lon, convert_fun=None, use_dem10=True):
51 base, lc, uc = _calc_from_latlon(lat, lon)
52
53 dem05fn = "__converted/DEM05/" + base + ".data"
54 dem10fn = "__converted/DEM10/" + base + ".data"
55
56 elevs1, elevs2 = None, None
57
58 if use_dem10 and os.path.exists(dem10fn):
59 elevs1 = _load0(dem10fn)
60
61 if os.path.exists(dem05fn):
62 elevs2 = _load0(dem05fn)
63
64 if elevs1 is None:
65 if elevs2 is None:
66 elevs = np.full((_shape[0], _shape[1]), np.nan)
67 print("data was not found: {}".format(base))
68 else:
69 elevs = elevs2
70 print("DEM5: {}".format(base))
71 else:
72 elevs = elevs1
73 if elevs2 is not None:
74 notnan = (np.isnan(elevs2) == False)
75 elevs[notnan] = elevs2[notnan]
76 print("DEM5 + DEM10: {}".format(base))
77 else:
78 print("DEM10: {}".format(base))
79
80 if convert_fun:
81 elevs = convert_fun(elevs)
82
83 return elevs, lc, uc
84
85
86 def _load_range0(lc_lat, lc_lon, uc_lat, uc_lon, convert_fun=None, use_dem10=True):
87 #
88 def _stfun(fun, left, right):
89 if left is None:
90 return right
91 return fun((left, right))
92 #
93 ydis = (2. / 3 / 8 / 10)
94 xdis = (1. / 8 / 10)
95
96 # adjust startpoint to center of mesh
97 # (for special boundary case.)
98 lc_lat = int(lc_lat / ydis) * ydis + ydis / 2.
99 lc_lon = int(lc_lon / xdis) * xdis + xdis / 2.
100
101 ycells = int(np.ceil(uc_lat / ydis) - np.floor(lc_lat / ydis))
102 xcells = int(np.ceil(uc_lon / xdis) - np.floor(lc_lon / xdis))
103 result, lc, uc = None, None, None
104 #
105 for i in range(ycells):
106 tmp = None
107 for j in range(xcells):
108 e, lc_tmp, uc_tmp = load(
109 lc_lat + ydis * i,
110 lc_lon + xdis * j,
111 convert_fun, use_dem10)
112 #
113 if not lc:
114 lc = lc_tmp
115 uc = uc_tmp
116 #
117 tmp = _stfun(np.hstack, tmp, e)
118 #
119 result = _stfun(np.vstack, result, tmp)
120 #
121 return result, lc, uc
122
123
124 def load_range(lat1, lon1, lat2, lon2, convert_fun=None, use_dem10=True):
125 lc_lat = min(lat1, lat2)
126 lc_lon = min(lon1, lon2)
127 uc_lat = max(lat1, lat2)
128 uc_lon = max(lon1, lon2)
129
130 elevs, lc, uc = _load_range0(
131 lc_lat, lc_lon, uc_lat, uc_lon,
132 convert_fun, use_dem10)
133 X = np.linspace(lc[1], uc[1], elevs.shape[1])
134 Y = np.linspace(lc[0], uc[0], elevs.shape[0])
135 xmin, xmax = np.where(X <= lc_lon)[0][-1], np.where(X >= uc_lon)[0][0]
136 ymin, ymax = np.where(Y <= lc_lat)[0][-1], np.where(Y >= uc_lat)[0][0]
137 return X[xmin:xmax], Y[ymin:ymax], elevs[ymin:ymax, xmin:xmax]
ハイライトした「notnan」部分は、「DEM10 をベースにし、DEM5 データの非NaN部分を取り込む」としていて、まぁこれがキモ。なんだけど、池などの「内水面」が DEM10 だと標高データ持ってるみたいで、ちょっと識別しにくくなるのが難点だな…。
なお、ほかのハイライト箇所2箇所、adjust startpoint to center of meshとある部分と np.where 部分はともに、最初から間違っていた境界処理の修正。これはこれまで書いてきた converted_loader.py 引用してる投稿全部に追記入れといた。(ぴったり境界位置だった場合に困ったことになっていた。)
以下結果。
上述房総半島について「DEM5のみ、DEM10と組み合わせ」をアニメーションGIFにしたもの:
おっけーだ。おっけーなんだけど、最大に拡大してじっくり見ると、解像度が変わる境界が実は線になって見えちゃったりすんのよね。まぁいいか…。
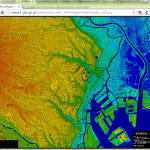
箱根・富士山(範囲「35.076203, 139.160385」~「35.539109, 138.540344」、zoom(1/3))は PDF でどーぞ:
Click here to download
Click here to download
解像度の変わり目が気になるかどうか、自分の目で確かめてみて。
にしても、「富士山だけ見たい」ってのがもう、かなりの広域扱わないといけないのね。山頂付近だけてならともかく、裾野からの全体をみたければ。上の絵にした領域だと、1/3ズームでも shape が (2777, 3720) になる。処理時間は3分30秒とかそんな。縮小しないと shape は (8331, 11160)。これはかなりキツいサイズ。(ちなみに最初の「関東平野全域」は、1/25 で 50分くらいかかってる。)