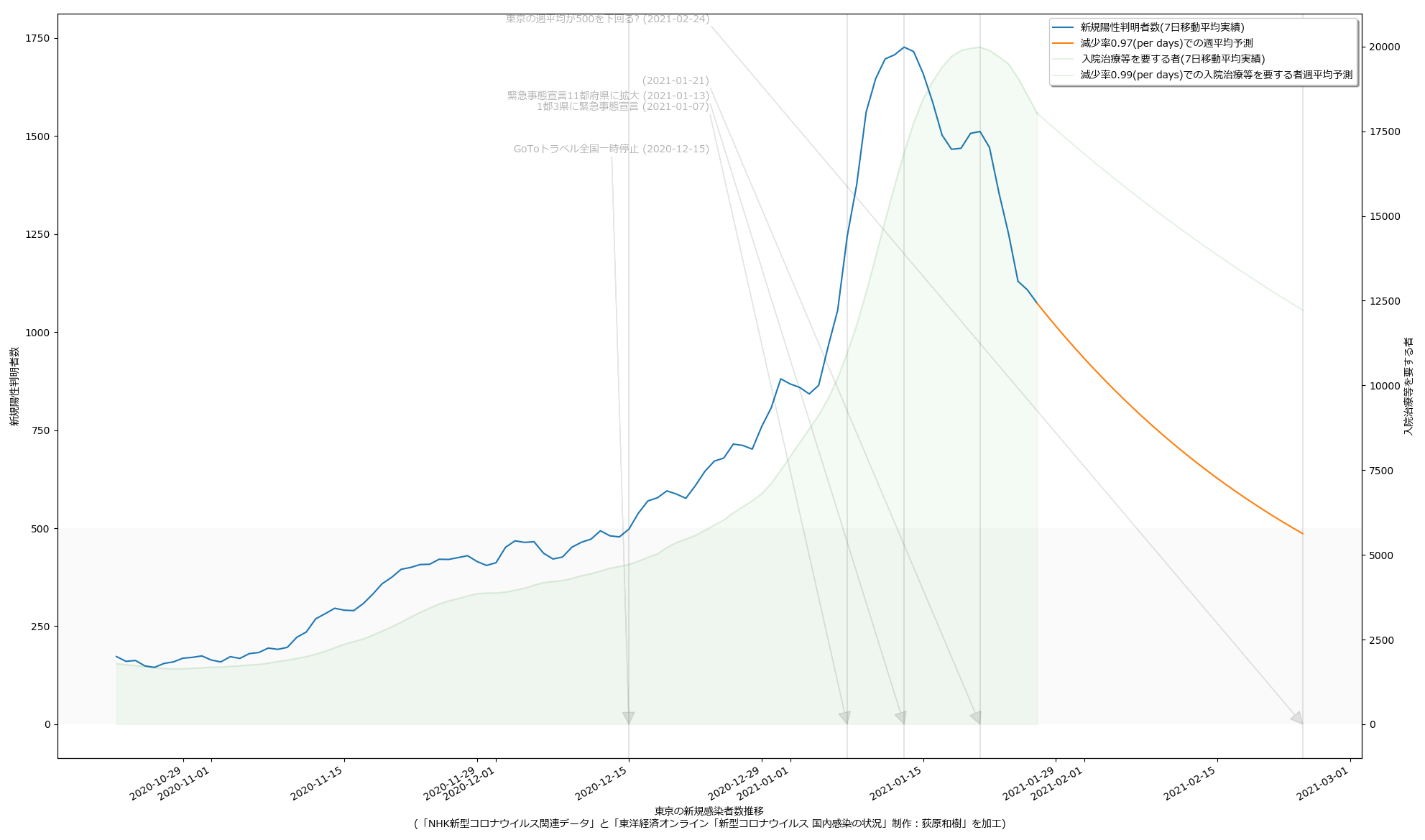
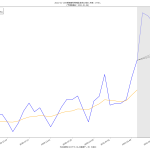
こういう可視化を共有すべきだ、てのが言いたいこと、てこと。
(8改)の追記に示したやつを、まぁ「見せれないこともない」くらいには整理したので:
1 # -*- coding: utf-8 -*-
2 import io
3 import sys
4 import csv
5 import datetime
6 import numpy as np
7
8
9 def _tonpa(fn):
10 # rownum=2020/1/16からの日数, colnum=都道府県コード-1
11 result = {
12 "newpat": np.zeros((1, 47)),
13 "newpatmovavg": np.zeros((1, 47)), # 週平均
14 "newpatmovavg2": np.zeros((1, 47)), # 2週平均
15 "newdeath": np.zeros((1, 47)),
16 "newdeathmovavg": np.zeros((1, 47)),
17 "newdeathmovavg2": np.zeros((1, 47)),
18 "effrepro": np.zeros((1, 48)), # 実効再生産数(47都道府県+全国)
19 }
20 era = "2020/1/16"
21 era = datetime.datetime(*map(int, era.split("/")))
22 reader = csv.reader(io.open(fn, encoding="utf-8-sig"))
23 next(reader)
24 for row in reader:
25 dt, (p, c, d) = row[0], map(int, row[1::2])
26 dt = (datetime.datetime(*map(int, dt.split("/"))) - era).days
27 if dt > len(result["newpat"]) - 1:
28 for k in result.keys():
29 cn = 48 if k == "effrepro" else 47
30 result[k] = np.vstack((result[k], np.zeros((1, cn))))
31 result["newpat"][dt, p - 1] = c
32 result["newpatmovavg"][dt, p - 1] = result[
33 "newpat"][max(0, dt - 7):dt + 1, p - 1].mean()
34 result["newpatmovavg2"][dt, p - 1] = result[
35 "newpat"][max(0, dt - 14):dt + 1, p - 1].mean()
36 result["newdeath"][dt, p - 1] = d
37 result["newdeathmovavg"][dt, p - 1] = result[
38 "newdeath"][max(0, dt - 7):dt + 1, p - 1].mean()
39 result["newdeathmovavg2"][dt, p - 1] = result[
40 "newdeath"][max(0, dt - 14):dt + 1, p - 1].mean()
41 # 実効再生産数は、現京都大学西浦教授提案による簡易計算(を一般向け説明にまとめてくれ
42 # ている東洋経済ONLINE)より:
43 # (直近7日間の新規陽性者数/その前7日間の新規陽性者数)^(平均世代時間/報告間隔)
44 # 平均世代時間は=5日、報告間隔=7日の仮定。
45 # のつもりだが、東洋経済ONLINEで GitHub にて公開(閉鎖予定)されてる計算結果の csv とは
46 # 値が違う。違うことそのものは致し方ないこと。計算タイミングによって結果は変わるし、
47 # NHK まとめと東洋経済ONLINEのデータはシンクロしてないから。思いっきり間違ってるって
48 # ことはなさそう。近い値にはなってる。
49 for dt in range(len(result["newpat"])):
50 # 都道府県別
51 sum1 = result["newpat"][dt-6:dt+1, :47].sum(axis=0)
52 sum2 = result["newpat"][dt-6-7:dt+1-7, :47].sum(axis=0)
53 result["effrepro"][dt, :47] = np.power(sum1 / sum2, 5. / 7.)
54
55 # 全国
56 sum1 = result["newpat"][dt-6:dt+1, :].sum()
57 sum2 = result["newpat"][dt-6-7:dt+1-7, :].sum()
58 result["effrepro"][dt, 47] = np.power(sum1 / sum2, 5. / 7.)
59 np.savez(io.open("nhk_news_covid19_prefectures_daily_data.npz", "wb"), **result)
60
61
62 if __name__ == '__main__':
63 fn = "nhk_news_covid19_prefectures_daily_data.csv"
64 _tonpa(fn)
スクリプトをマジメに読んだ人は気付いたかもしれない。「8日平均」と「15日平均」を計算しちゃってる。一連の「主張」の本題とはあまり関係ないとはいえ、さすがにもうちょっと慎重にやるべきでした、すまぬ。正しくは「dt – 6」「dt – 5」…「dt – 1」「dt」の7エレメントでの平均を取らなければならないので、「slice(dt – 6, dt + 1)」。8日平均が役に立たないわけではないけれど、やりたいこととやってることが違うので、間違いは間違い。グラフは滑らかさが若干減って、もうほんの少しだけガタガタになる。
1 # -*- coding: utf-8-unix -*-
2 import io
3 import csv
4 import datetime
5 import numpy as np
6
7
8 _PREFCODE = {
9 '北海道': 1,
10 '青森県': 2,
11 '岩手県': 3,
12 '宮城県': 4,
13 '秋田県': 5,
14 '山形県': 6,
15 '福島県': 7,
16 '茨城県': 8,
17 '栃木県': 9,
18 '群馬県': 10,
19 '埼玉県': 11,
20 '千葉県': 12,
21 '東京都': 13,
22 '神奈川県': 14,
23 '新潟県': 15,
24 '富山県': 16,
25 '石川県': 17,
26 '福井県': 18,
27 '山梨県': 19,
28 '長野県': 20,
29 '岐阜県': 21,
30 '静岡県': 22,
31 '愛知県': 23,
32 '三重県': 24,
33 '滋賀県': 25,
34 '京都府': 26,
35 '大阪府': 27,
36 '兵庫県': 28,
37 '奈良県': 29,
38 '和歌山県': 30,
39 '鳥取県': 31,
40 '島根県': 32,
41 '岡山県': 33,
42 '広島県': 34,
43 '山口県': 35,
44 '徳島県': 36,
45 '香川県': 37,
46 '愛媛県': 38,
47 '高知県': 39,
48 '福岡県': 40,
49 '佐賀県': 41,
50 '長崎県': 42,
51 '熊本県': 43,
52 '大分県': 44,
53 '宮崎県': 45,
54 '鹿児島県': 46,
55 '沖縄県': 47,
56 }
57 _COLSNM = (
58 "testedPositive",
59 "peopleTested",
60 "hospitalized",
61 "serious",
62 "discharged",
63 "deaths",
64 "effectiveReproductionNumber",
65
66 "hospitalized_movavg"
67 )
68 def _tonpa(fn):
69 # 元データ prefectures.csv の著作権:
70 # 『東洋経済オンライン「新型コロナウイルス 国内感染の状況」制作:荻原和樹』
71 #
72 # 1. 開始日が NHK まとめと違う
73 # 2. 更新のない都道府県は省略してある
74 # 3. 日付は一応抜けなく連続している、今のところ
75 #
76 # NHKまとめのほうで密行列として扱った同じノリにしたい、ここでは面倒だけど。
77 # つまり、列方向に都道府県、行方向に日付。
78 result = {
79 k: np.zeros((1, 47))
80 for k in _COLSNM}
81 reader = csv.reader(io.open(fn, encoding="utf-8-sig"))
82 next(reader)
83 def _f(d):
84 if not d or d == "-":
85 # 不明とか未報告の意味だと思う。つまりこれはゼロではないので、
86 # 可搬性のためには NaN の方が本当はベターだけれど、ここではあまり考えないでおく。
87 return 0
88 return float(d)
89
90 era = datetime.datetime(2020, 1, 16) # NHKまとめのほうの起点日
91 curdate = era
92 for line in reader:
93 date = datetime.datetime(*map(int, line[:3]))
94 dadv = (date - curdate).days
95 data = list(map(_f, line[5:]))
96 p = _PREFCODE[line[3]]
97 for i, k in enumerate(_COLSNM):
98 for _ in range(dadv):
99 result[k] = np.vstack((result[k], np.zeros((1, 47))))
100 if i < len(_COLSNM) - 1:
101 result[k][-1, p - 1] = data[i]
102 dt = (date - era).days
103 result["hospitalized_movavg"][dt, p - 1] = result[
104 "hospitalized"][max(0, dt - 7):dt + 1, p - 1].mean()
105 curdate = date
106 np.savez(io.open("toyokeizai_online_covid19.npz", "wb"), **result)
107
108
109 if __name__ == '__main__':
110 _tonpa("covid19-master/data/prefectures.csv")
GitHub での公開をあさってやめてしまう、って、すごーくイヤなタイミングなのだが、その後もデータ公開そのものは行われるとのことなので、期待せずに期待しておこう…。
1 # -*- coding: utf-8 -*-
2 import io
3 import datetime
4 import functools
5 import numpy as np
6 from scipy.optimize import curve_fit
7 import matplotlib as mpl
8 import matplotlib.pyplot as plt
9 import matplotlib.font_manager
10
11
12 def _vis(dat, dattko):
13 fontprop = matplotlib.font_manager.FontProperties(
14 fname="c:/Windows/Fonts/meiryo.ttc")
15
16 era = "2020/1/16" # 木曜日
17 era = datetime.datetime(*map(int, era.split("/")))
18 (wh_sl, wh_n) = (slice(12, 12+1), "東京")
19
20 npat = dat["newpat"][:,wh_sl].sum(axis=1)
21 npatma = dat["newpatmovavg"][:,wh_sl].sum(axis=1)
22 #hosp = dattko["hospitalized"][:,wh_sl].sum(axis=1)
23 hospma = dattko["hospitalized_movavg"][:,wh_sl].sum(axis=1)
24 if len(npat) > len(hospma):
25 # 東洋経済のほうが更新が一日遅い。
26 npat = npat[:-(len(npat) - len(hospma))]
27 npatma = npatma[:-(len(npatma) - len(hospma))]
28
29 T = [era + datetime.timedelta(t) for t in range(len(npat))]
30 fig, ax1 = plt.subplots(tight_layout=True)
31 fig.set_size_inches(16.53 * 1.2, 11.69)
32 ax2 = ax1.twinx()
33 skip = 7*4*10
34 #
35 # ワイドショーが最近やってる「単純計算でこのまま~%減少ペースならば云々」そのもの
36 # を。簡単のために直近の最大(1月13日)と本日の比較とする。計算は要は「a^t * n0」の
37 # a を求める問題。
38 #
39 n0 = npatma[skip:].max()
40 [t0], = np.where(npatma[skip:] == n0) # 1月13日
41 def _fcurve(r, t, a):
42 return r * np.power(a, t)
43 t1 = len(npatma[skip:]) - 1
44 popt, pcov = curve_fit(
45 functools.partial(_fcurve, (n0,)),
46 np.arange(t1 - t0 + 1), npatma[skip:][t0:])
47 preds = []
48 val = npatma[skip:][t1]
49 while True:
50 preds.append(val)
51 if val < 500.: # 東京の感染者数週平均が500を下回る?
52 break
53 val *= popt[0]
54 #
55 lines = []
56 l, = ax1.plot(T[skip:], npatma[skip:])
57 lines.append(l)
58 l, = ax1.plot(
59 [era + datetime.timedelta(skip + t)
60 for t in range(t1, t1 + len(preds))], preds)
61 lines.append(l)
62 #
63 l, = ax2.plot(T[skip:], hospma[skip:], "g", alpha=0.1)
64 ax2.fill_between(T[skip:], hospma[skip:], alpha=0.05, color="tab:green")
65 lines.append(l)
66 #
67 hn0 = hospma[skip:].max()
68 [hmax], = np.where(hospma[skip:] == hn0)
69 popt2, pcov2 = curve_fit(
70 functools.partial(_fcurve, (hn0,)),
71 np.arange(t1 - hmax + 1), hospma[skip:][hmax:])
72 l, = ax2.plot(
73 [era + datetime.timedelta(skip + t)
74 for t in range(t1, t1 + len(preds))],
75 [_fcurve(hospma[skip:][t1], t - t1, popt2[0])
76 for t in range(t1, t1 + len(preds))],
77 "g", alpha=0.1)
78 lines.append(l)
79 #
80 ax1.legend(
81 lines,
82 ["新規陽性判明者数(7日移動平均実績)",
83 "減少率{:.2f}(per days)での週平均予測".format(popt[0]),
84 "入院治療等を要する者(7日移動平均実績)",
85 "減少率{:.2f}(per days)での入院治療等を要する者週平均予測".format(popt2[0]),
86 ],
87 shadow=True, prop=fontprop)
88 ax1.set_xlabel(
89 """{}の新規感染者数推移
90 (「NHK新型コロナウイルス関連データ」と「東洋経済オンライン「新型コロナウイルス 国内感染の状況」制作:荻原和樹」を加工)""".format(
91 wh_n),
92 fontproperties=fontprop)
93 ax1.set_ylabel("新規陽性判明者数", fontproperties=fontprop)
94 ax2.set_ylabel("入院治療等を要する者", fontproperties=fontprop)
95 ax1.axhspan(0, 500, facecolor="lightgray", alpha=0.1)
96 fig.autofmt_xdate()
97 #
98 tlast = era + datetime.timedelta(skip + t1 + len(preds) - 1)
99 #
100 for evtxt, evdt in (
101 ("GoToトラベル全国一時停止", datetime.datetime(2020, 12, 15)),
102 ("1都3県に緊急事態宣言", datetime.datetime(2021, 1, 7)),
103 ("緊急事態宣言11都府県に拡大", datetime.datetime(2021, 1, 13)),
104 ("東京の週平均が500を下回る?", tlast),
105 ("", T[skip + hmax]),
106 ):
107 ax1.annotate("{} ({})".format(evtxt, str(evdt)[:len("????-??-??")]),
108 xy=(evdt, 0), xycoords='data',
109 xytext=(
110 0.5,
111 (evdt - era).days / (tlast - era).days),
112 textcoords='axes fraction',
113 arrowprops=dict(facecolor='black', width=0.0001, alpha=0.1),
114 horizontalalignment='right', verticalalignment='top',
115 alpha=0.3,
116 fontproperties=fontprop)
117 ax1.vlines([evdt], 0, 1, transform=ax1.get_xaxis_transform(), alpha=0.1)
118 #
119 #plt.show()
120 fig.savefig(
121 "nhk_news_covid19_prefectures_daily_data5_2_4.png")
122 plt.close(fig)
123
124
125 if __name__ == '__main__':
126 fn1 = "nhk_news_covid19_prefectures_daily_data.npz"
127 fn2 = "toyokeizai_online_covid19.npz"
128 _vis(np.load(io.open(fn1, "rb")), np.load(io.open(fn2, "rb")))
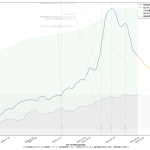
改めて主張を整理しておく。「ワタシの予測をみよ」てことではない、てことを強調しとく。正直「精度が良いとは思えない素人予測」そのものはかなりどうでもいい。
政治家「ども」が「根拠なき楽観論に立ってしまう」もしくは「正常性バイアスにかかる」ことの理由の一つは、そもそも政治家に限らず「未来予測に対するコンセンサスが形成されていないから」である。専門家の正しい意見よりも世論にビクつくのが今の政権のようだから、「一般市民が共通の未来予測をみる」ことで世論を動かせば、「遅すぎる対応」の緩和は、メディアの力を通じて可能なのではないのか、ということ。
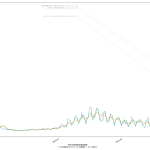
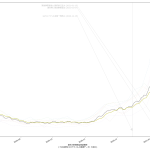
ゆえに、「簡単でもいいから一週間後~二週間後くらいまでの近未来予測を出せ」(そしてメディア同士で競え)というのが一つ。もう一つが、「新規感染者だけをみようとするから見誤る」の件。「入院治療等を要する者」を皆で共有する方が、たぶん医療逼迫度合いは伝わりやすいのではないか、てこと。可視化してみせたように、ピークアウトしていると思われる日付は、「入院治療等を要する者」の方が一週間遅れている。両方毎日みるのがいいと思うし、そもそも「今日は新規が1000人でしたきゃーやべー」ではねくて、「本日時点で「感染状態扱い」の数が1万8000人である」と伝えられた方が、よりヤバさが伝わるんではないのか、てことね。そして(8改)の追記で書いた通り、本当はもう一歩進めてこの「感染状態扱い」の中の「入院/宿泊療養/自宅療養」も同時に可視化すべきだろう、てことね。
そして、そういう図を毎日一般市民が目にするようになれば、ワタシは政府の動きは最低でも一週間は早く動けるようになるんではないかと思ってる。何度も言うけど「同じ世界をみてない」ことが問題なんだよ。そしてそういうのを補っていくのが本来メディアの役目。
ところで「新規感染者が500人を下回る」タイミングで「入院治療等を要する者」が減ってもまだ12500人いる、としたら、これはかなりショッキングな気がするよね。どうなんだろうか。
2021-01-30 01:40追記:
今時刻時点でダウンロードできる最新データで更新:
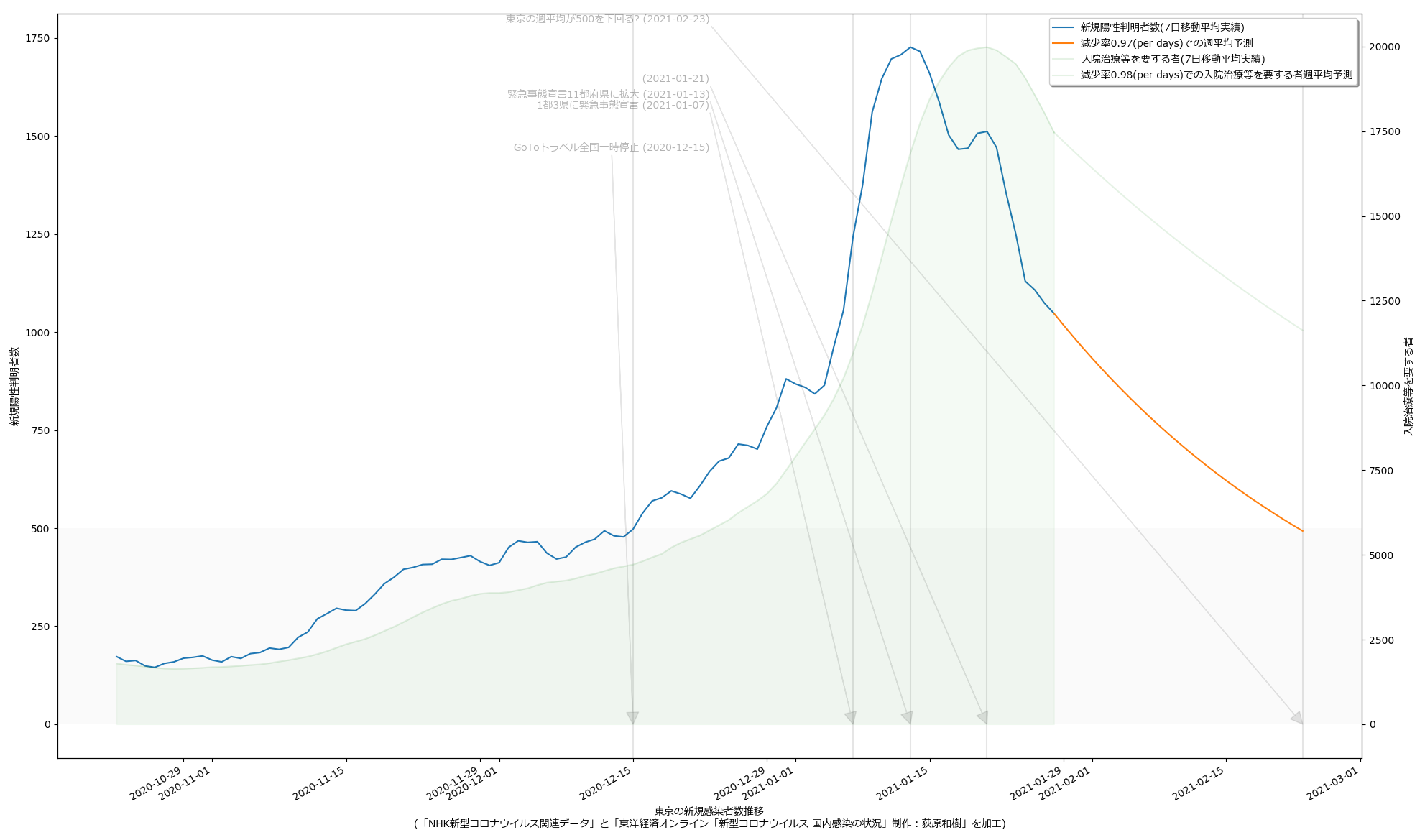
ワタシのアホ予測で少し減率がアップしている。
ところで、ワタシのアホ予測でなくて、今朝大学の先生がワタシのより遥かに早く減少する予測を立ててた。2/7に520人くらい、とかだったかしら。もっと減ってたっけ? 鵜吞みには出来ないなとは思ったけれど、予測に使っているのは西浦さんのと同じ数理モデルなんだって。少なくとも一般人のものとは違って「いい加減なものではない」ので、信じてみるのもいい。
結局ね、「議論」が「異なる未来予測の戦い」であるならいいわけよ、そうなって欲しいの。けど、今の「分断」は、結局のところ「未来予測と「思考停止」の戦い」だから皆イラついてるわけだ。(あと未来志向でないだけでなくて、他人事だから、だよな。)