1 # -*- coding: utf-8 -*-
2 import io
3 import sys
4 import csv
5 import datetime
6 import numpy as np
7
8
9 def _tonpa(fn):
10 # rownum=2020/1/16からの日数, colnum=都道府県コード-1
11 result = {
12 "newpat": np.zeros((1, 47)),
13 "newpatmovavg": np.zeros((1, 47)), # 週平均
14 "newpatmovavg2": np.zeros((1, 47)), # 2週平均
15 "newdeath": np.zeros((1, 47)),
16 "newdeathmovavg": np.zeros((1, 47)),
17 "newdeathmovavg2": np.zeros((1, 47)),
18 "effrepro": np.zeros((1, 48)), # 実効再生産数(47都道府県+全国)
19 }
20 era = "2020/1/16"
21 era = datetime.datetime(*map(int, era.split("/")))
22 reader = csv.reader(io.open(fn, encoding="utf-8-sig"))
23 next(reader)
24 for row in reader:
25 dt, (p, c, d) = row[0], map(int, row[1::2])
26 dt = (datetime.datetime(*map(int, dt.split("/"))) - era).days
27 if dt > len(result["newpat"]) - 1:
28 for k in result.keys():
29 cn = 48 if k == "effrepro" else 47
30 result[k] = np.vstack((result[k], np.zeros((1, cn))))
31 result["newpat"][dt, p - 1] = c
32 result["newpatmovavg"][dt, p - 1] = result[
33 "newpat"][max(0, dt - 7):dt + 1, p - 1].mean()
34 result["newpatmovavg2"][dt, p - 1] = result[
35 "newpat"][max(0, dt - 14):dt + 1, p - 1].mean()
36 result["newdeath"][dt, p - 1] = d
37 result["newdeathmovavg"][dt, p - 1] = result[
38 "newdeath"][max(0, dt - 7):dt + 1, p - 1].mean()
39 result["newdeathmovavg2"][dt, p - 1] = result[
40 "newdeath"][max(0, dt - 14):dt + 1, p - 1].mean()
41 # 実効再生産数は、現京都大学西浦教授提案による簡易計算(を一般向け説明にまとめてくれ
42 # ている東洋経済ONLINE)より:
43 # (直近7日間の新規陽性者数/その前7日間の新規陽性者数)^(平均世代時間/報告間隔)
44 # 平均世代時間は=5日、報告間隔=7日の仮定。
45 # のつもりだが、東洋経済ONLINEで GitHub にて公開(閉鎖予定)されてる計算結果の csv とは
46 # 値が違う。違うことそのものは致し方ないこと。計算タイミングによって結果は変わるし、
47 # NHK まとめと東洋経済ONLINEのデータはシンクロしてないから。思いっきり間違ってるって
48 # ことはなさそう。近い値にはなってる。
49 for dt in range(len(result["newpat"])):
50 # 都道府県別
51 sum1 = result["newpat"][dt-6:dt+1, :47].sum(axis=0)
52 sum2 = result["newpat"][dt-6-7:dt+1-7, :47].sum(axis=0)
53 result["effrepro"][dt, :47] = np.power(sum1 / sum2, 5. / 7.)
54
55 # 全国
56 sum1 = result["newpat"][dt-6:dt+1, :].sum()
57 sum2 = result["newpat"][dt-6-7:dt+1-7, :].sum()
58 result["effrepro"][dt, 47] = np.power(sum1 / sum2, 5. / 7.)
59 np.savez(io.open("nhk_news_covid19_prefectures_daily_data.npz", "wb"), **result)
60
61
62 if __name__ == '__main__':
63 fn = "nhk_news_covid19_prefectures_daily_data.csv"
64 _tonpa(fn)
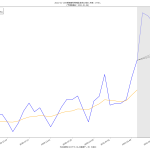
スクリプトをマジメに読んだ人は気付いたかもしれない。「8日平均」と「15日平均」を計算しちゃってる。一連の「主張」の本題とはあまり関係ないとはいえ、さすがにもうちょっと慎重にやるべきでした、すまぬ。正しくは「dt – 6」「dt – 5」…「dt – 1」「dt」の7エレメントでの平均を取らなければならないので、「slice(dt – 6, dt + 1)」。8日平均が役に立たないわけではないけれど、やりたいこととやってることが違うので、間違いは間違い。グラフは滑らかさが若干減って、もうほんの少しだけガタガタになる。
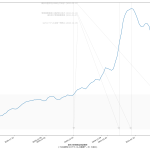
感染速度が「東京では鈍化しているようにみえる」と思うわけなのだが、「東京だけみてると見誤らないかしら」とも一方で感じていて。だからといって:
1 # -*- coding: utf-8 -*-
2 import io
3 import datetime
4 import numpy as np
5 import matplotlib as mpl
6 import matplotlib.pyplot as plt
7 import matplotlib.font_manager
8
9
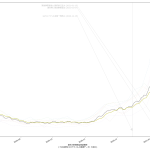
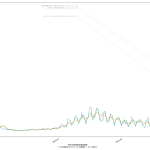
10 def _vis(dat):
11 fontprop = matplotlib.font_manager.FontProperties(
12 fname="c:/Windows/Fonts/meiryo.ttc")
13
14 era = "2020/1/16" # 木曜日
15 era = datetime.datetime(*map(int, era.split("/")))
16 # 11: '埼玉県'
17 # 12: '千葉県'
18 # 13: '東京都'
19 # 14: '神奈川県'
20 for i, (wh_sl, wh_n) in enumerate((
21 (slice(12, 12+1), "東京"),
22 # 2021-04-23: ゴメン、↓の slice、間違ってる。slice(10, 13+1) が正しい。
23 (slice(11, 14+1), "埼玉、千葉、東京、神奈川"))):
24 npat = dat["newpat"][:,wh_sl].sum(axis=1)
25 npatma = dat["newpatmovavg"][:,wh_sl].sum(axis=1)
26 npatma2 = dat["newpatmovavg2"][:,wh_sl].sum(axis=1)
27
28 T = [era + datetime.timedelta(t) for t in range(len(npat))]
29 fig, ax1 = plt.subplots(tight_layout=True)
30 fig.set_size_inches(16.53, 11.69)
31 skip = 7*4*10
32 lines = []
33 for dow in range(7):
34 l, = ax1.plot(
35 T[skip:][dow::7], npat[skip:][dow::7], linestyle="dotted", alpha=0.2)
36 lines.append(l)
37 l, = ax1.plot(T[skip:], npatma[skip:])
38 lines.append(l)
39 l, = ax1.plot(T[skip:], npatma2[skip:])
40 lines.append(l)
41 ax1.legend(lines, [
42 dow + "曜日" for dow in [
43 "木", "金", "土", "日", "月", "火", "水"]] + ["週平均", "2週平均"],
44 shadow=True, prop=fontprop)
45 ax1.set_xlabel(
46 "{}の新規感染者数推移\n(「NHK新型コロナウイルス関連データ」を加工)".format(
47 wh_n),
48 fontproperties=fontprop)
49 fig.autofmt_xdate()
50 #
51 for evtxt, evdt in (
52 ("GoToトラベル全国一時停止", datetime.datetime(2020, 12, 15)),
53 ("1都3県に緊急事態宣言", datetime.datetime(2021, 1, 7)),
54 ("緊急事態宣言11都府県に拡大", datetime.datetime(2021, 1, 13)),
55 ):
56 ax1.annotate("{} ({})".format(evtxt, str(evdt)[:len("????-??-??")]),
57 xy=(evdt, 0), xycoords='data',
58 xytext=(
59 0.5,
60 (evdt - era).days / (T[-1] - era).days),
61 textcoords='axes fraction',
62 arrowprops=dict(facecolor='black', width=0.0001, alpha=0.1),
63 horizontalalignment='right', verticalalignment='top',
64 alpha=0.3,
65 fontproperties=fontprop)
66 ax1.vlines([evdt], 0, 1, transform=ax1.get_xaxis_transform(), alpha=0.1)
67 #
68 #plt.show()
69 fig.savefig(
70 "nhk_news_covid19_prefectures_daily_data2_{}.png".format(
71 i))
72 plt.close(fig)
73
74
75 if __name__ == '__main__':
76 fn = "nhk_news_covid19_prefectures_daily_data.npz"
77 _vis(np.load(io.open(fn, "rb")))
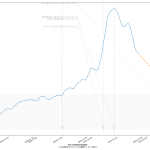
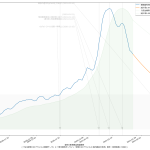
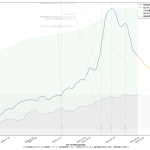
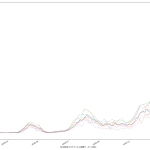
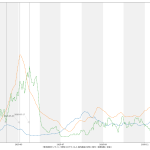
としてみたが、人口比まで加味しないと結局東京をみてるのと大差ないのかも:
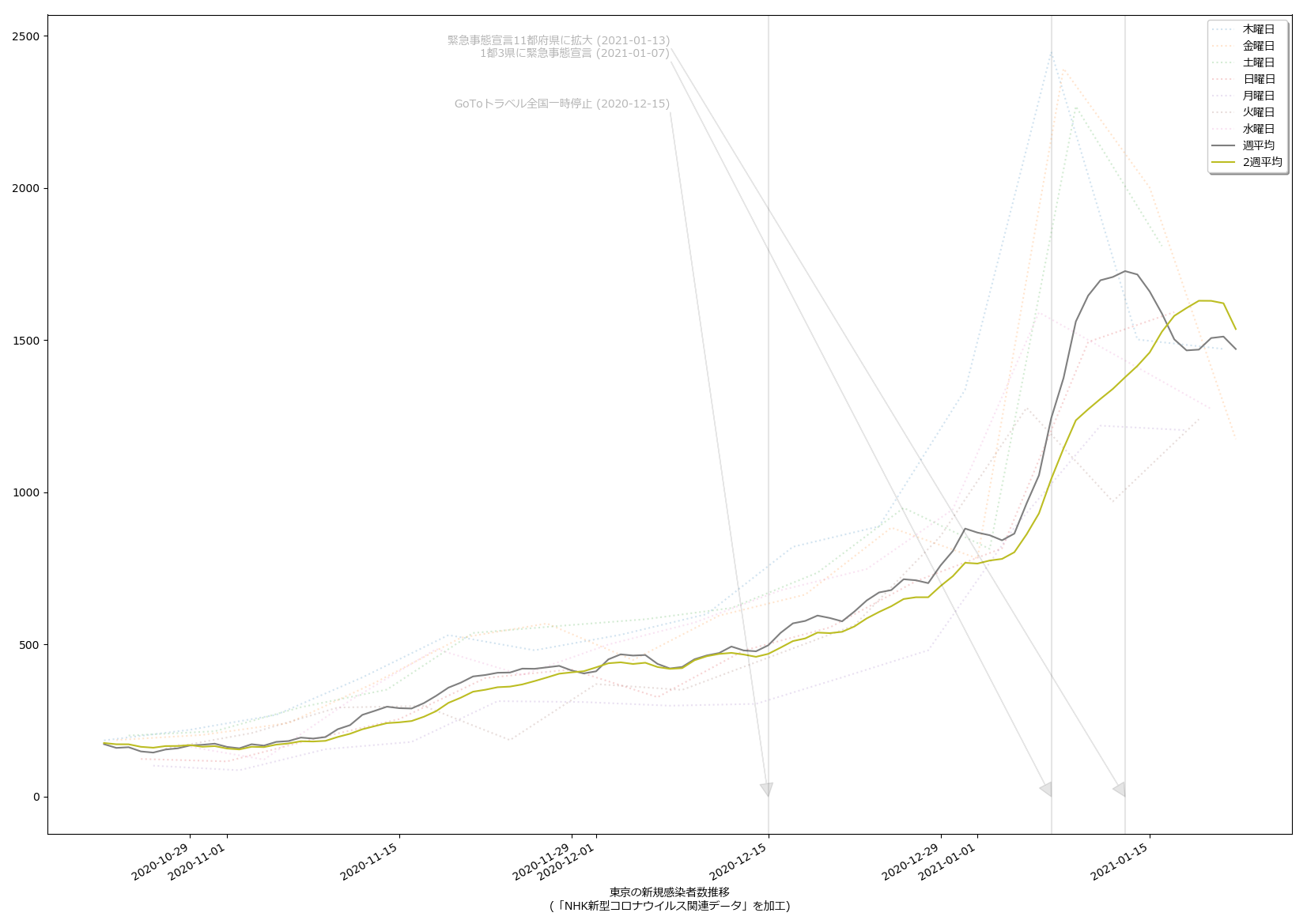
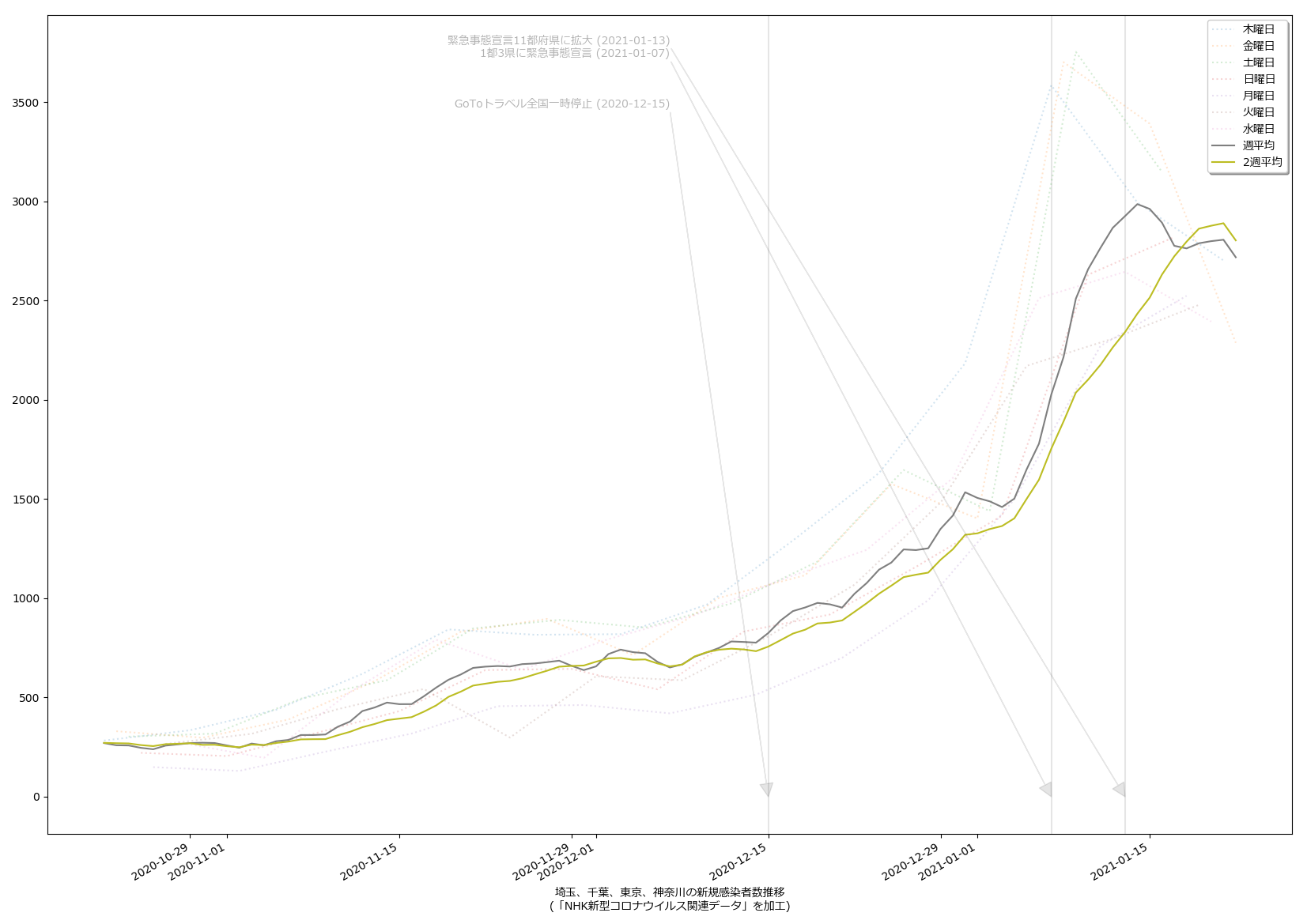
ただ、東京だけをみてるのよりも、ピーク位置などが遅れて出てるので、そうね、この見方はより安全かもしらんね。
こうしてみてみた感じとしては、少なくとも生の感染者数だけでみた場合、一都三県としても感染速度は鈍化してる、とみてよさそうだなとは思った。
ちなみに、(5)の話の続きをしとく。「岡田理論(?)」な二週間平均もようやく落ち始めた、ので、「大丈夫」なのかい? ほらね、そういうことを言わなければならなくなる。結局「本日」を終端としてひくグラフというのは、あーーーーーーーーーーっっっったりまえのこととして「過去の履歴」なのだから、「下降してた」ことだけは言えて、「いや、下降してない」というのが言い方としての間違い。そうじゃなくて、「下降傾向にあるとは言えないので本日以降に下がるとは言い切れない」という「未来志向」の言い方を意識してしないと、少なくともマジメに考える人には滅茶苦茶違和感になるし、マジメに考えない人には「反感」を与えると思う。
あと、ニュースやワイドショーでの伝え方にはやっぱり言いたいんだけれど、どうにも「収束予測」の際にしか未来予想をしないのはどうにかすべき。昨日だったかので、「~%の減率ならば~月~日に500人を下回る」という予測をやってるのがあった。なんでこれを「このままいくとヤバい」警鐘のために日々伝えようという発想にならんのだろう。いまだに「今日1000人、きゃーびっくりびっくり」と大差ないことを繰り返してる。さすがにバカなの死ぬの、と言いたくはなる。